Capítulo 4 Análise do fatorial 2k sem repetições
Quando o número de fatores \(k\) é grande, o modelo contém muitos parâmetros e o experimento muitos pontos experimentais. Fazer repetições dos pontos experimentais, embora seja interessante porque fornece uma estimativa pura da variância do erro, \(r\)-plica o número de unidades experimentais. Isso pode facilmente inviabilizar a investigação.
Em muitos contexto, pode-se considerar o princípio da esparsidade que diz que o sistema é geralmente dominado pelos efeitos principais e interações de ordem baixa. As interações de ordem alta, e a medida que mais altas forem, contribuem menos, ao ponto de, a partir de certo grau, poderem ser negligenciadas (MONTGOMERY; RUNGER, 2009). Colocando de outra forma, para prever o resposta de um dado sistema ou fenômeno, a maior contribuição é proviniente dos efeitos principais, seguidos das interações duplas, e assim por diante. Dessa forma, os termos de alto podem ser vistos como estimadores do variância do erro.
A realização de experimentos fatoriais \(2^k\) é feita baseada nesse princípio. Como o número de parâmetros do modelo é \(p = 2^k\), não restam graus de liberdade (ou desvios livres) para obter uma estimativa pura da variância do erro. Ou seja, o modelo é saturado e ajusta-se perfeitamente aos dados não produzindo desvios entre valores observados e ajustados. Sem uma estivativa para a variância do erro, não é possível testar o efeito dos termos no modelo pelo quadro de análise de variância. Aí é que se usa o princípio da esparsidade.
Para obter uma estimativa de variância do erro, abandona-se os termos de maior ordem do modelo. Pode-se a interação de ordem \(k\). Ou as de ordem \(k\) e \(k-1\). Ou ainda as de ordem \(k\) até \(k - 2\) conforme seja o valor de \(k\). Essa estimativa de variância não é pura mas sim proveniente do abandono de termos de alta ordem que considera-se não ter efeito. Sob a hipótese de não haver efeito desses termos, o quadrado médio de cada um deles é um estimador independente da variância do erro. Recorde-se das esperanças matemáticas dos quadrados médios, \(\text{E}(QM)\). A questão em aberto é: até que grau de interação pode-se abondonar para compor uma estimativa da variância do erro?
Uma forma visual de decidir sobre os graus a serem abandonados é usar o gráfico quantil-quantil normal (qq-normal) com as estimativas dos efeitos. Qual a razão para isso? É a seguinte. Sob a hipótese nula, que estabelece não haver efeito de nenhum termo do modelo, os efeitos estimados (exceto intercepto) são variáveis aleatórias com média 0 e variância \(\sigma^2/(r 2^k)\) (no caso, \(r = 1\)). Terem a mesma variância deve-se ao uso da escala codificada das variáveis. Com isso, se não houver efeito dos termos, o gráfico qq-normal não irá indicar afastamento da normalidade. Quando houver algum afastamento, é porque o efeito de algum termo é diferente de zero. Então é só etiquetar os pontos no gráfico e verificar o grau daquele conjunto de pontos que está mais próximo de zero para devidir o grau dos termos ou até mesmo os termos que serão abandonados do modelo para compor a variância do erro.
É possível fazer simulações sob a hipótese nula para criar uma banda de confiança para o gráfico que auxilie determinar os efeitos mais salientes. O gráfico com bandas ou envelope é bastante usado como instrumento de diagnóstico em modelo de regressão. No entanto, existem várias formas de determinar as bandas. Umas por reamostragem dos dados observados, outras por simulação sob a hipótese nula, etc. Esta última é a que faz sentido para o contexto. Mais sobre as diferenças de abordagem para construção das bandas são vistas em CE 089 · Estatística Computacional II.
O fragmento de código a seguir constrói o gráfico qq-normal e adiciona bandas de confiança, provenientes do intervalo de confiança quantílico para as estatísticas de ordem sob hipótese nula.
par(mfrow = c(1, 2))
# Simulando efeitos de um fatorial 2^4 sob hipótese nula e uma variância
# qualquer conhecida para os efeitos, no caso Var(ef) = 1.
ef <- rnorm(2^4 - 1, mean = 0, sd = 1)
# Gráfico qq-normal.
qqnorm(ef)
qqline(ef)
# Simula sob a hipótese nula.
L <- replicate(n = 2999,
expr = {
# Retorna a amostra ordenada.
sort(rnorm(2^4 - 1, mean = 0, sd = 1))
})
# Determina intervalos de confiança quantílicos para cada esatística de
# ordem.
ic <- apply(cbind(L, sort(ef)),
MARGIN = 1,
FUN = quantile,
probs = c(0.025, 0.5, 0.975))
# Gráfico do observado contra o resultado sob hipótese nula.
qq <- qqnorm(ef)
abline(h = 0, lty = 3, col = "gray")
matlines(x = sort(qq$x), y = t(ic),
type = "l", lty = 2, col = 1)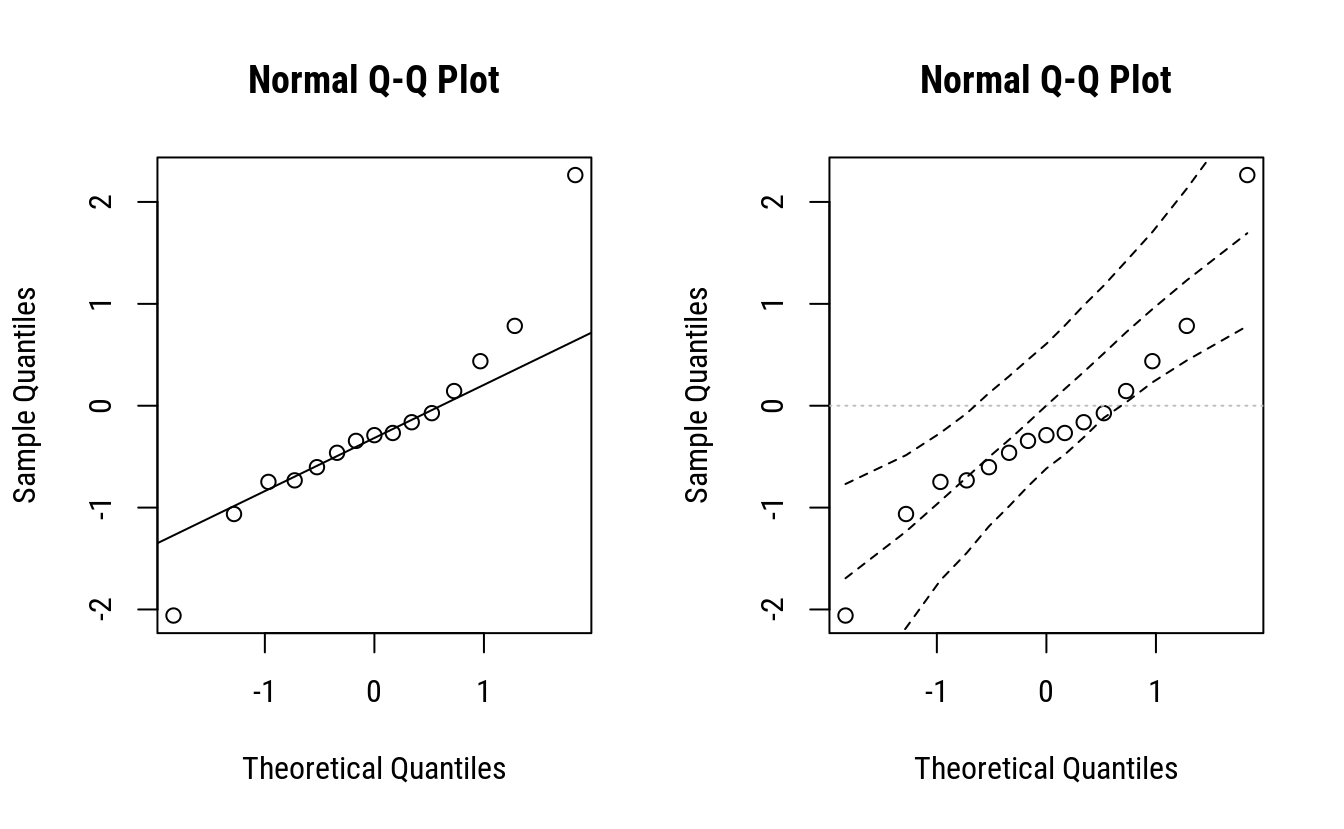
layout(1)O gráfico qq-normal com envelope seria uma opção interessante, porém, não tem-se como determinar o evelope porque não se conhece a variância dos efeitos. Na simulação foi considerado como 1. Na prática a variância dos efeitos é \(\sigma^2/(r 2^k)\). O valor de \(r = 1\) e \(k\) é conhecido. Mas o valor de \(\sigma^2\) não é conhecido e não se dispõe de uma estivativa para ele. Afinal, o que está sendo discutido é justamente quais o termos abandonar para obter-se uma estimativa para \(\sigma^2\). Portanto, usa-se o gráfico qq-normal sem o evelope porque não se tem todas as informações para construí-lo.
A decisão sobre quais os termos ou graus abadonar será pela disposição dos termos no gráfico. Aqueles efeitos com estimativas mais próximas de zero são os candidatos naturais para serem abadonados.
Depois que um certo número de termos for abandonado, será possível testar a significância dos termos mantidos pelo quadro de análise de variância. O fato dos efeitos serem ortogonais faz com que os efeitos associados aos termos mantidos não precisem ser reestimados devido ao abandono dos demais.
Nos estudos de caso a seguir serão analisados dados de experimentos fatoriais \(2^k\) sem réplicas para os pontos experimentais.
library(tidyverse)
library(lattice)4.1 Rendimento de processo químico
Um planejamento fatorial \(2^4\) foi corrido em um processo químico. Os fatores do planejamento são A = tempo, B = concentração, C = pressão, D = temperatura. A variável resposta é rendimento.
Esses dados são do exerício 14-20, na página 355 do MONTGOMERY; RUNGER (2009).
4.1.1 Análise exploratória
Nessa seção serão feitos os cálculos para obter as estimativas dos parâmetros, somas de quadrados, predição e erros padrões. Na seção seguinte os dados serão analisados com funções do R e será feita discussão dos resultados.
rm(list = objects())
l <- c(-1, 1)
ex14.20 <- expand.grid(A = l, B = l, C = l, D = l,
KEEP.OUT.ATTRS = FALSE)
# ex14.20$y <- scan()
# dput(ex14.20$y)
ex14.20$y <- c(12, 18, 13, 16, 17, 15, 20, 15, 10, 25, 13, 24, 19, 21,
17, 23)
my_plot <- function(f1, f2, y, data) {
ggplot(data = data.frame(x = data[[f1]],
color = data[[f2]],
y = data[[y]]),
mapping = aes(x = x,
y = y,
color = factor(color),
group = color)) +
geom_point() +
stat_summary(geom = "line", fun.y = "mean") +
labs(x = f1, y = y, color = f2)
}
# Gráficos para relações mariginais de 2 fatores.
gridExtra::grid.arrange(
ncol = 2,
my_plot("A", "B", "y", ex14.20),
my_plot("A", "C", "y", ex14.20),
my_plot("B", "C", "y", ex14.20),
my_plot("A", "D", "y", ex14.20),
my_plot("B", "D", "y", ex14.20),
my_plot("C", "D", "y", ex14.20))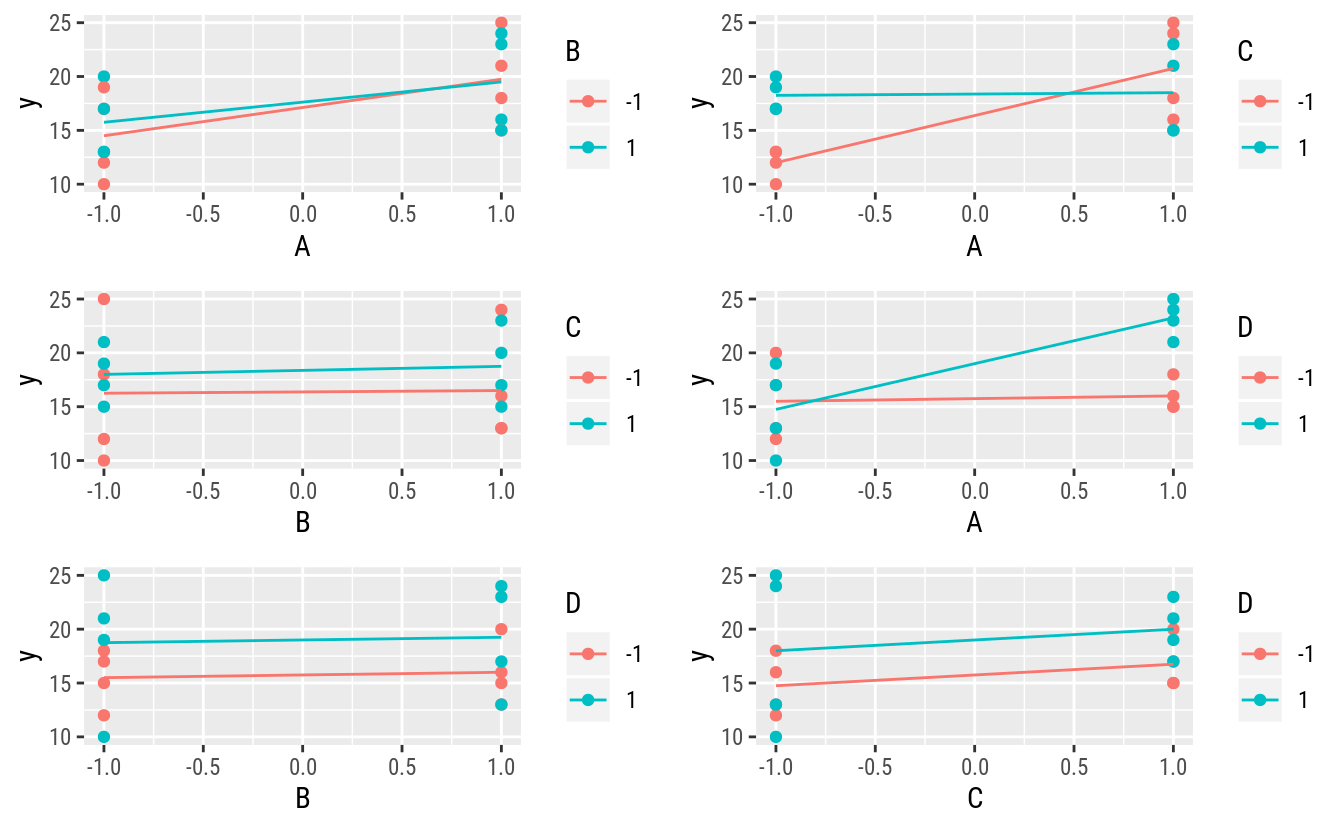
Os gráficos mostram um indicativo de efeito de A ou talvez interação de A com C e de A com D. Não parece haver efeito de B.
4.1.2 Análise feita de forma operacional
# Matriz do modelo e vetor resposta.
# X <- model.matrix(~A * B * C * D, data = ex14.20)
X <- with(ex14.20,
cbind(I = 1,
A, B, C, D,
AB = A * B,
AC = A * C,
BC = B * C,
AD = A * D,
BD = B * D,
CD = C * D,
ABC = A * B * C,
ABD = A * B * D,
ACD = A * C * D,
BCD = B * C * D,
ABCD = A * B * C * D))
y <- cbind(ex14.20$y)
# Matriz do modelo ao lado do vetor da resposta.
data.frame(X, "|" = "|", y, check.names = FALSE)## I A B C D AB AC BC AD BD CD ABC ABD ACD BCD ABCD | y
## 1 1 -1 -1 -1 -1 1 1 1 1 1 1 -1 -1 -1 -1 1 | 12
## 2 1 1 -1 -1 -1 -1 -1 1 -1 1 1 1 1 1 -1 -1 | 18
## 3 1 -1 1 -1 -1 -1 1 -1 1 -1 1 1 1 -1 1 -1 | 13
## 4 1 1 1 -1 -1 1 -1 -1 -1 -1 1 -1 -1 1 1 1 | 16
## 5 1 -1 -1 1 -1 1 -1 -1 1 1 -1 1 -1 1 1 -1 | 17
## 6 1 1 -1 1 -1 -1 1 -1 -1 1 -1 -1 1 -1 1 1 | 15
## 7 1 -1 1 1 -1 -1 -1 1 1 -1 -1 -1 1 1 -1 1 | 20
## 8 1 1 1 1 -1 1 1 1 -1 -1 -1 1 -1 -1 -1 -1 | 15
## 9 1 -1 -1 -1 1 1 1 1 -1 -1 -1 -1 1 1 1 -1 | 10
## 10 1 1 -1 -1 1 -1 -1 1 1 -1 -1 1 -1 -1 1 1 | 25
## 11 1 -1 1 -1 1 -1 1 -1 -1 1 -1 1 -1 1 -1 1 | 13
## 12 1 1 1 -1 1 1 -1 -1 1 1 -1 -1 1 -1 -1 -1 | 24
## 13 1 -1 -1 1 1 1 -1 -1 -1 -1 1 1 1 -1 -1 1 | 19
## 14 1 1 -1 1 1 -1 1 -1 1 -1 1 -1 -1 1 -1 -1 | 21
## 15 1 -1 1 1 1 -1 -1 1 -1 1 1 -1 -1 -1 1 -1 | 17
## 16 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 | 23k <- 4 # Número de fatores.
r <- 1 # Número de repetições.
# Contrates são o principal artefato. É apenas X'y.
ctr <- crossprod(X, y)
ctr## [,1]
## I 278
## A 36
## B 4
## C 16
## D 26
## AB -6
## AC -34
## BC 2
## AD 32
## BD 0
## CD 0
## ABC 8
## ABD 6
## ACD -2
## BCD -6
## ABCD 8# Estimativas dos parâmetros.
# beta <- solve(crossprod(X), crossprod(X, y))
beta <- diag(1/(r * 2^k), 2^k) %*% ctr
beta## [,1]
## [1,] 17.375
## [2,] 2.250
## [3,] 0.250
## [4,] 1.000
## [5,] 1.625
## [6,] -0.375
## [7,] -2.125
## [8,] 0.125
## [9,] 2.000
## [10,] 0.000
## [11,] 0.000
## [12,] 0.500
## [13,] 0.375
## [14,] -0.125
## [15,] -0.375
## [16,] 0.500# Estimativa dos parâmetros (por somatórios basicamente).
ctr/(r * 2^k) # idem ao `beta`## [,1]
## I 17.375
## A 2.250
## B 0.250
## C 1.000
## D 1.625
## AB -0.375
## AC -2.125
## BC 0.125
## AD 2.000
## BD 0.000
## CD 0.000
## ABC 0.500
## ABD 0.375
## ACD -0.125
## BCD -0.375
## ABCD 0.500# Somas de quadrados.
tail(ctr, n = -1)^2/(r * 2^k)## [,1]
## A 81.00
## B 1.00
## C 16.00
## D 42.25
## AB 2.25
## AC 72.25
## BC 0.25
## AD 64.00
## BD 0.00
## CD 0.00
## ABC 4.00
## ABD 2.25
## ACD 0.25
## BCD 2.25
## ABCD 4.00# Valores ajustados para cada ponto experimental.
ex14.20$hy <- X %*% beta
# Resíduos.
ex14.20$res <- with(ex14.20, y - hy)
c(ex14.20$res) # ATTENTION: os resíduos são zero!## [1] 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0# Desvio padrão residual.
s2 <- with(ex14.20, sum(res^2)/(nrow(X) - ncol(X)))
s2## [1] NaN# NOTE: não tem como estimar a variância residual (pura) porque não tem
# repetições.
# Gráfico quantil-quantil normal das estimativas.
qq <- qqnorm(c(beta)[-1], pch = 19, col = "orange")
qqline(c(beta)[-1], lty = 2)
text(x = qq$x, y = qq$y,
labels = colnames(X)[-1],
pos = ifelse(qq$x < 0, 4, 2))
rect(-1.04, -0.49, 0.61, 0.63, lty = 2, border = "red")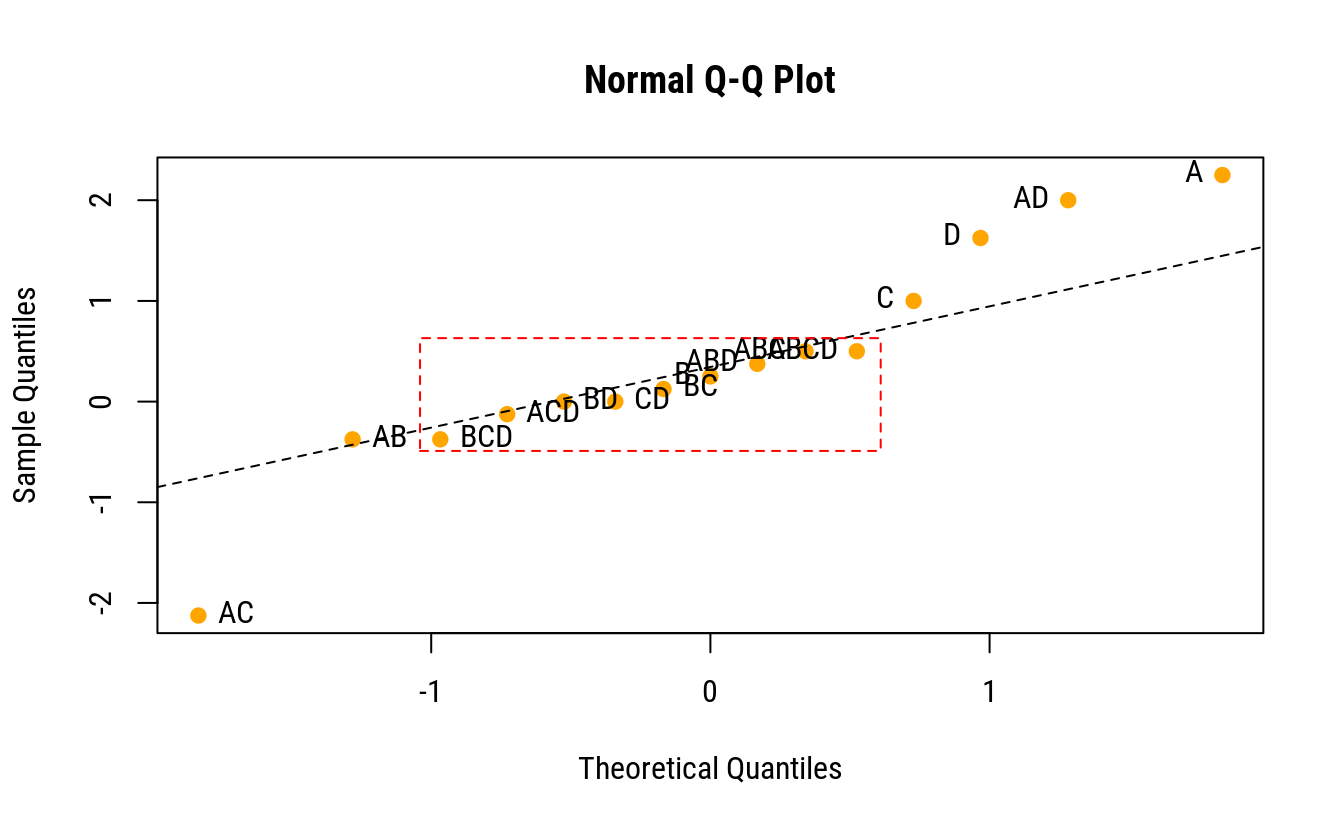
# Manterndo termos de interação até a dupla.
j <- nchar(colnames(X)) <= 2
X1 <- X[, j]
beta1 <- beta[j, ]
# Valores ajustados com o modelo reduzido.
ex14.20$hy <- X1 %*% beta1
# Resíduos.
ex14.20$res <- with(ex14.20, y - hy)
c(ex14.20$res)## [1] 0.125 0.625 0.125 -0.875 -0.875 0.125 0.625 0.125 -1.125
## [10] 0.375 0.875 -0.125 1.875 -1.125 -1.625 0.875# Desvio padrão residual.
s2 <- with(ex14.20, sum(res^2)/(nrow(X1) - ncol(X1)))
s2## [1] 2.554.1.3 Análise feita com funções
Na lm() declaramos o modelo contendos todos os termos possível, ou seja, todos os termos até a interação de ordem \(k = 4\). Com isso serão estimados \(2^4\) e tem-se \(2^k\) observações já que os pontos experimentais não possuem repetição. É sabido que esse modelo irá se ajustar perfeitamente aos dados e não se terá uma estimativa da variância do erro para testar os efeitos.
# Ajuste do modelo saturado (consome todos os graus de liberdade).
m0 <- lm(y ~ A * B * C * D, data = ex14.20)
# NOTE: não é possível ver gráfico dos resíduos porque todos os resíduos
# são 0.
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 81.00 81.00
## B 1 1.00 1.00
## C 1 16.00 16.00
## D 1 42.25 42.25
## A:B 1 2.25 2.25
## A:C 1 72.25 72.25
## B:C 1 0.25 0.25
## A:D 1 64.00 64.00
## B:D 1 0.00 0.00
## C:D 1 0.00 0.00
## A:B:C 1 4.00 4.00
## A:B:D 1 2.25 2.25
## A:C:D 1 0.25 0.25
## B:C:D 1 2.25 2.25
## A:B:C:D 1 4.00 4.00
## Residuals 0 0.00Conforme comentado, o quadro de análise de variância contém as somas de quadrados porém, a soma de quadrados dos resíduos é zero. Com isso não pode obter a estatística F para testar o efeito dos termos do modelo. A abordagem do gráfico qq-normal será usada para indicar refinamentos no modelo.
cfs <- coef(m0)[-1] %>%
enframe()
# Gráfico quantil-quantil normal das estimativas.
qq <- qqnorm(cfs$value, pch = 19, col = "orange")
text(x = qq$x, y = qq$y,
labels = cfs$name,
pos = ifelse(qq$x < 0, 4, 2))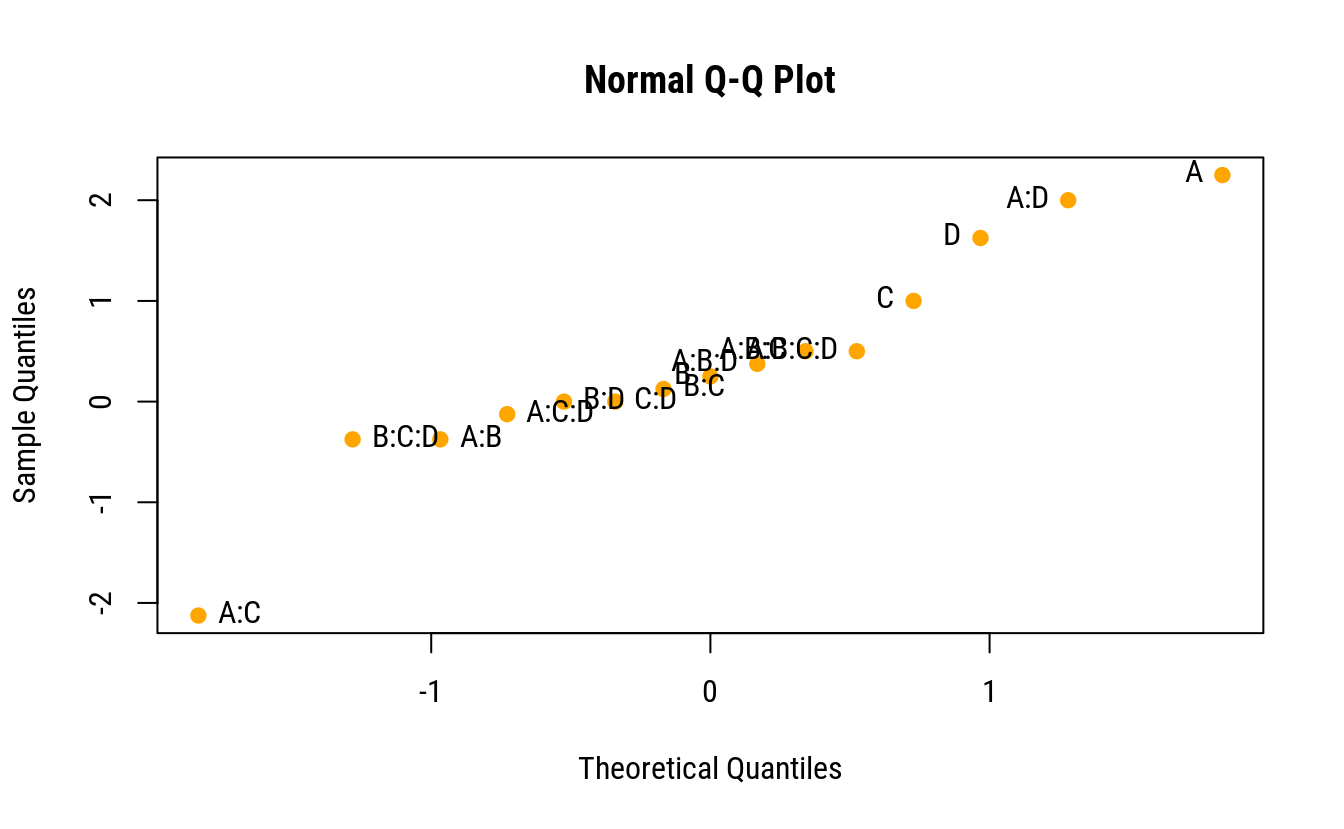
ggplot(data = cfs,
mapping = aes(x = reorder(name, value), y = value)) +
geom_segment(aes(xend = name, yend = 0)) +
geom_segment(data = data.frame(x = 3.5, xend = 11.5, y = -0.25),
color = "orange",
arrow = arrow(length = unit(0.1, "inches"),
angle = 90,
ends = "both"),
mapping = aes(x = x, y = y, xend = xend, yend = y))
# Gráfico quantil-quantil normal nos efeitos estimados.
# FrF2::DanielPlot(m0, pch = 19)Pela análise do gráfico QQ-normal, os termos mais próximo de zero incluem as interações de 4 e 3 ordem. Pode-se então, para ter uma estimativa de variância residual, abandonar esses termos do modelo. Essa estimativa não é uma estimativa pura de variância residual mas sim inteiramente proveniente de termos omitidos do modelo.
Os dois modelos a seguir são refinamentos consecutivos do modelo inicial. O primeiro considera todos os efeitos principais e as interações duplas com o fator A. O segundo, baseado no quadro de ANOVA do primeiro, abandona o termos do fator B que não se mostrou relevante.
# Ajuste do modelo reduzido, com alguns termos de até 2 grau.
m1 <- update(m0, . ~ A * (B + C + D))
anova(m1)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 81.00 81.000 49.8462 0.0001061 ***
## B 1 1.00 1.000 0.6154 0.4553663
## C 1 16.00 16.000 9.8462 0.0138499 *
## D 1 42.25 42.250 26.0000 0.0009310 ***
## A:B 1 2.25 2.250 1.3846 0.2731392
## A:C 1 72.25 72.250 44.4615 0.0001578 ***
## A:D 1 64.00 64.000 39.3846 0.0002390 ***
## Residuals 8 13.00 1.625
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Mais reduzido ainda com a remoção de B.
m2 <- update(m0, . ~ A * (C + D))
anova(m2)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 81.00 81.000 49.8462 3.456e-05 ***
## C 1 16.00 16.000 9.8462 0.0105485 *
## D 1 42.25 42.250 26.0000 0.0004647 ***
## A:C 1 72.25 72.250 44.4615 5.583e-05 ***
## A:D 1 64.00 64.000 39.3846 9.193e-05 ***
## Residuals 10 16.25 1.625
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Gráficos serão usados para estudar o efeito dos fatores no comportamento da resposta. Uma malha fina nos níveis dos fatores é gerada e o valor predito é calculado para cada ponto. Assim, gráficos de contornos ou superfície podem ser usados para exibir a relação entre os fatores.
Como a interação AC tem maior efeito que AD, optou-se por fazer os gráficos de superfície considerando esse par de fatores para cada nível fixado do fator D. Todavia, outras escolhas sejam igualmente interessantes de inspecionar. Não necessariamente a escolha tem que ser baseada no tamanho do efeito. É importante que haja sentido prático em estudá-la ou compreendê-la.
# Malha de valores para predição.
pred <- expand.grid(A = seq(-1, 1, by = 0.1),
C = seq(-1, 1, by = 0.1),
D = seq(-1, 1, by = 0.5))
pred <- cbind(pred,
as.data.frame(predict(m2,
newdata = pred,
se.fit = TRUE)[1:2]))
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = A, y = C, z = fit, fill = fit)) +
facet_wrap(facets = ~D) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black") +
coord_equal()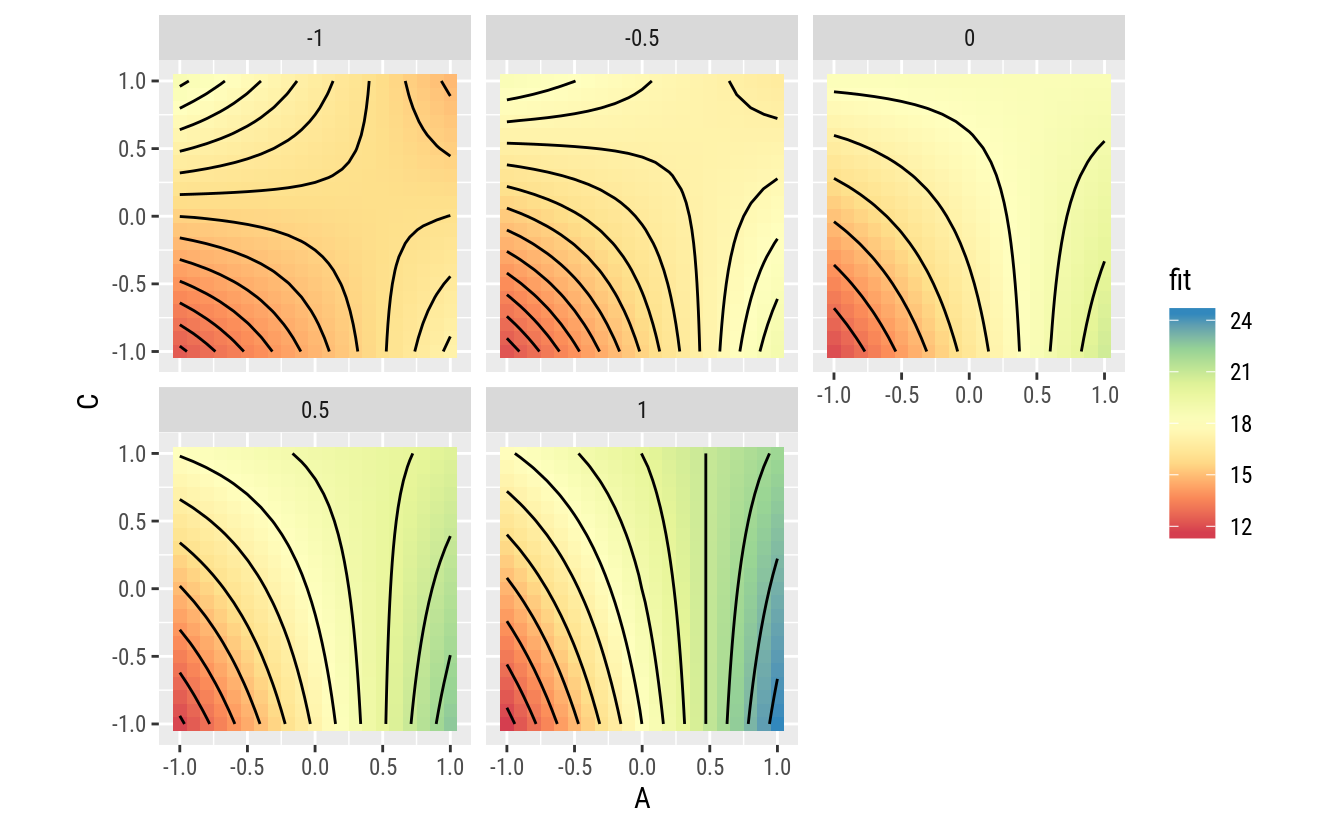
# Gráficos de linhas.
ggplot(data = pred,
mapping = aes(x = A, y = fit, color = C, group = C)) +
facet_wrap(facets = ~D) +
geom_line() +
scale_color_distiller(palette = "BrBG")
Os gráficos indicam bem a existência de interação de A com cada um dos fatores C e D. Para um mesmo nível de D, as linhas que descrevem o efeito de A para níveis de C mudam de inclinação com a variação de C. Para um mesmo nível de C (linha de mesma cor), a inclinação que representa o efeito de A muda com os níveis de D.
Os gráfico a seguir exibe a relação em uma superfície 3D.
# Esquema de cores para o gráfico com a `lattice`.
colr <- RColorBrewer::brewer.pal(11, "Spectral")
colr <- colorRampPalette(colr, space = "rgb")
# Adicionando os contornos sobre a superfície. Requer funções externas.
source(paste0("https://raw.githubusercontent.com/walmes/wzRfun/",
"master/R/panel.3d.contour.R"))
# Gráfico de superfície em 3D com isolinhas (ainda não recomendo).
wireframe(fit ~ A + C | factor(D),
data = pred,
strip = strip.custom(strip.names = c(TRUE, TRUE),
var.name = "D"),
as.table = TRUE,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100))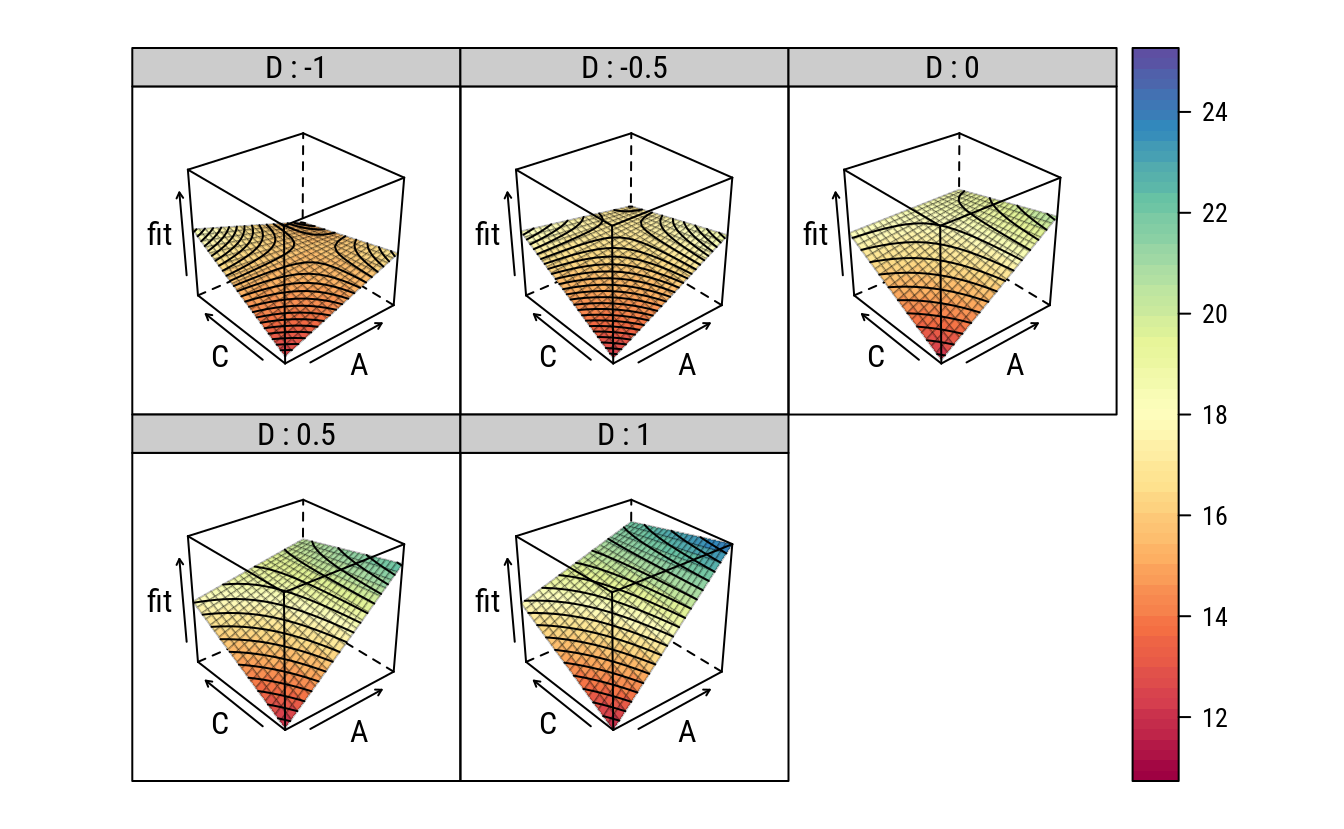
# Valores preditos em cada ponto experimental.
grid <- unique(ex14.20[, c("A", "C", "D")])
grid$fit <- predict(m2, newdata = grid)
arrange(grid, fit)## A C D fit
## 1 -1 -1 1 11.625
## 2 -1 -1 -1 12.375
## 3 1 1 -1 14.875
## 4 1 -1 -1 17.125
## 5 -1 1 1 17.875
## 6 -1 1 -1 18.625
## 7 1 1 1 22.125
## 8 1 -1 1 24.375Dentro da região experimental, sob os pontos de suporte, o maior valor predito para a resposta está sob a condição de operação \(\{1, -1, 1\}\) em A, C e D, respectivamente. Isso significa então que a o rendimento é maior para um nível alto de tempo (A), nível baixo de pressão (C) e nível alto de temperatura (D). Esse é portanto, a condição de ótimo local, restrito à região experimental. Questiona-se se não haveriam condições experimentais além da região atual que fossem ainda melhores.
É possível extrapolar, a partir o modelo ajustado, e predizer a resposta para condições de operação além daquelas investigadas. Por outro lado, essas previsões que são extrapolações podem estar equivocadas e somente novos experimentos feitos na região de interesse que irão dar uma resposta segura sobre o redimento da reação. Pode-se definir uma nova região experimental a partir da direção de maior inclinação, considerada direção mais promissora, para encontrar o máximo do rendimento com base no modelo que foi ajustado.
4.2 Resistência à compressão do concreto
Os dados mostrados a seguir representam uma única réplica de um planejamento \(2^5\) que é usado em um experimento para estudar a resistência à compressão do concreto. Os fatores são mistura (A), tempo (B), laboratório (C), temperatura (D) e tempo de secagem (E).
Esses dados são do exerício 14-14, na página 354 do MONTGOMERY; RUNGER (2009).
# Criação dos dados.
l <- c(-1, 1)
ex14.14 <- expand.grid(A = l, B = l, C = l, D = l, E = l,
KEEP.OUT.ATTRS = FALSE)
# ex14.14$y <- scan()
# dput(ex14.14$y)
ex14.14$y <- c(7, 9, 34, 55, 6, 10, 30, 53, 10, 11, 30, 61, 8, 11, 33,
60, 8, 12, 35, 62, 6, 12, 30, 55, 19, 15, 40, 65, 15, 20,
34, 68)
str(ex14.14)## 'data.frame': 32 obs. of 6 variables:
## $ A: num -1 1 -1 1 -1 1 -1 1 -1 1 ...
## $ B: num -1 -1 1 1 -1 -1 1 1 -1 -1 ...
## $ C: num -1 -1 -1 -1 1 1 1 1 -1 -1 ...
## $ D: num -1 -1 -1 -1 -1 -1 -1 -1 1 1 ...
## $ E: num -1 -1 -1 -1 -1 -1 -1 -1 -1 -1 ...
## $ y: num 7 9 34 55 6 10 30 53 10 11 ...my_plot <- function(f1, f2, y, data) {
ggplot(data = data.frame(x = data[[f1]],
color = data[[f2]],
y = data[[y]]),
mapping = aes(x = x,
y = y,
color = factor(color),
group = color)) +
geom_point() +
stat_summary(geom = "line", fun.y = "mean") +
labs(x = f1, y = y, color = f2)
}
# Alguns para relações mariginais de 2 fatores.
gridExtra::grid.arrange(
ncol = 2,
my_plot("A", "B", "y", ex14.14),
my_plot("B", "A", "y", ex14.14),
my_plot("D", "E", "y", ex14.14),
my_plot("E", "D", "y", ex14.14))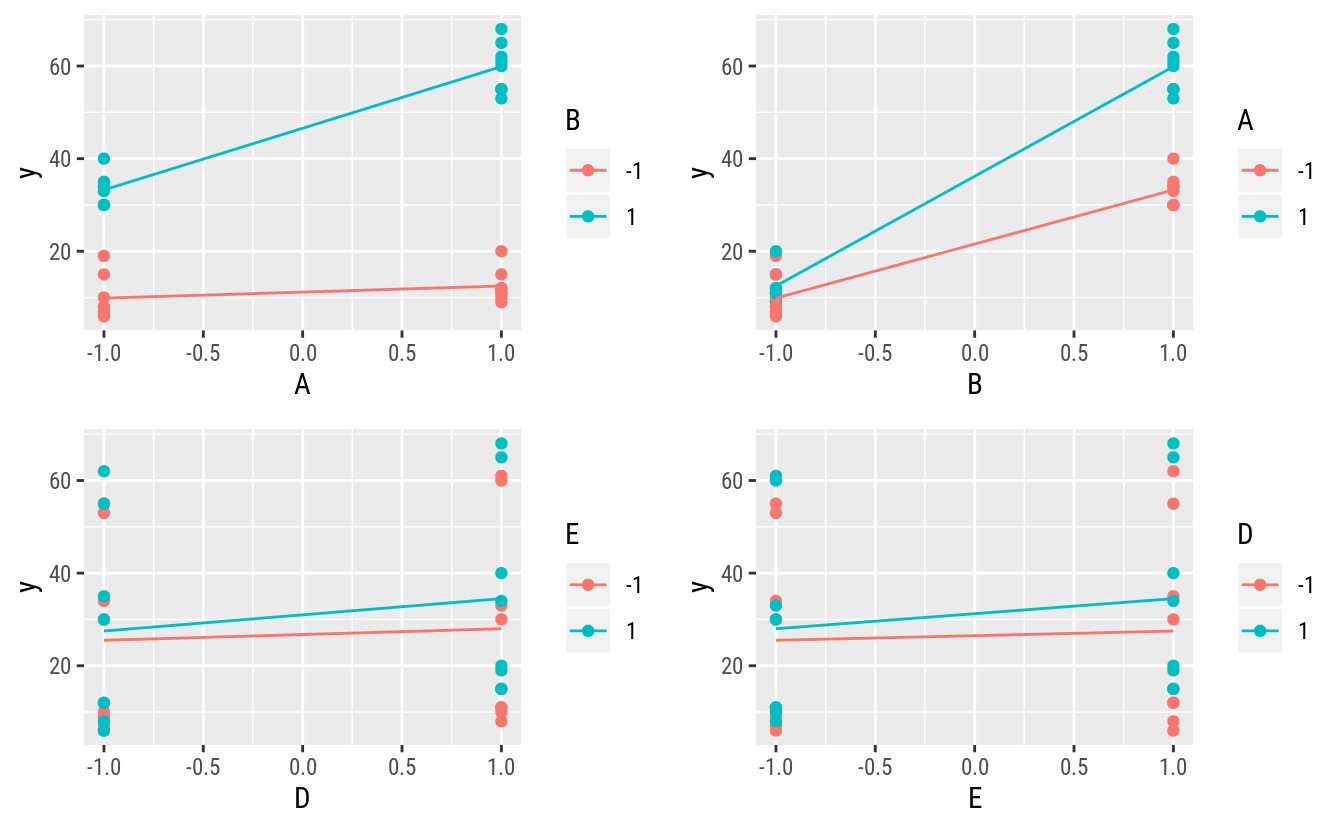
4.2.1 Análise feita de forma operacional
Veja o código disponível na primeira seção. Apenas é necessário fazer a adaptação para o número de fatores.
4.2.2 Análise feita com funções
# Ajuste do modelo saturado (consome todos os graus de liberdade).
m0 <- lm(y ~ A * B * C * D * E, data = ex14.14)
# NOTE: não é possível ver gráfico dos resíduos porque todos os resíduos
# são 0.
# # Quadro de análise de variância.
# anova(m0)
cfs <- coef(m0)[-1] %>%
enframe()
# Gráfico quantil-quantil normal das estimativas.
qq <- qqnorm(cfs$value, pch = 19, col = "orange")
qqline(cfs$value, lty = 2)
text(x = qq$x, y = qq$y,
labels = cfs$name,
pos = ifelse(qq$x < 0, 4, 2))
rect(-1.29, -0.852, 0.994, 1.515, lty = 2, border = "red")
ggplot(data = cfs,
mapping = aes(x = reorder(name, value), y = value)) +
geom_segment(aes(xend = name, yend = 0)) +
geom_segment(data = data.frame(x = 2.5, xend = 25.5, y = 2),
color = "orange",
arrow = arrow(length = unit(0.1, "inches"),
angle = 90,
ends = "both"),
mapping = aes(x = x, y = y, xend = xend, yend = y)) +
theme(axis.text.x = element_text(angle = 90, hjust = 1, vjust = 0.5))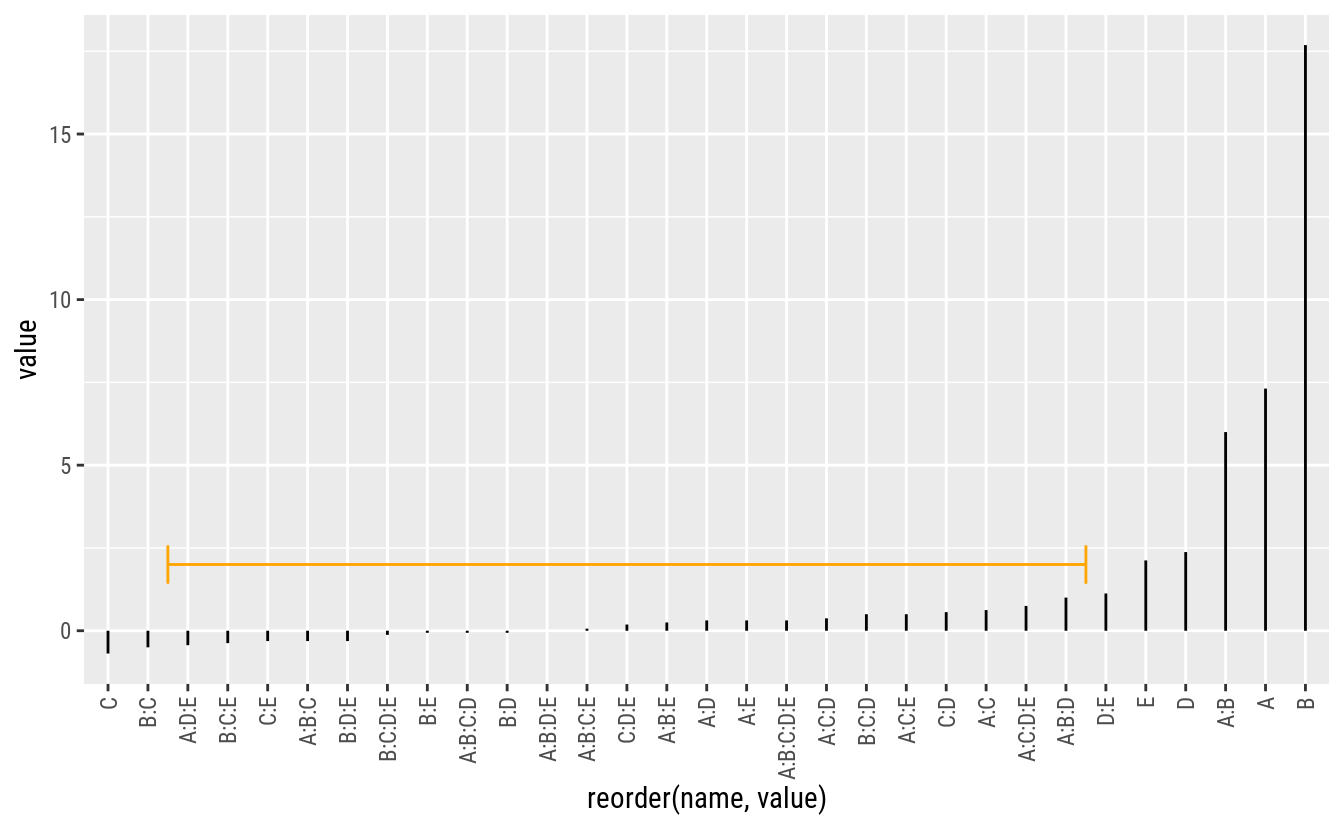
# Gráfico quantil-quantil normal nos efeitos estimados.
# FrF2::DanielPlot(m0, pch = 19)Pela análise do gráfico QQ-normal, os termos mais próximo de zero incluem as interações acima de 3 ordem. Pode-se então, para ter uma estimativa de variância residual, abandonar esses termos do modelo. Essa estimativa não é uma estimativa pura de variância residual mas sim inteiramente proveniente de termos omitidos do modelo.
# Ajuste do modelo reduzido com termos de 3 grau.
m1 <- update(m0, . ~ (A + B + C + D + E)^3)
anova(m1)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 1711.1 1711.1 469.3371 6.315e-07 ***
## B 1 10011.1 10011.1 2745.9086 3.242e-09 ***
## C 1 15.1 15.1 4.1486 0.0878322 .
## D 1 180.5 180.5 49.5086 0.0004118 ***
## E 1 144.5 144.5 39.6343 0.0007483 ***
## A:B 1 1152.0 1152.0 315.9771 2.037e-06 ***
## A:C 1 12.5 12.5 3.4286 0.1135317
## A:D 1 3.1 3.1 0.8571 0.3902585
## A:E 1 3.1 3.1 0.8571 0.3902585
## B:C 1 8.0 8.0 2.1943 0.1890296
## B:D 1 0.1 0.1 0.0343 0.8592010
## B:E 1 0.1 0.1 0.0343 0.8592010
## C:D 1 10.1 10.1 2.7771 0.1466681
## C:E 1 3.1 3.1 0.8571 0.3902585
## D:E 1 40.5 40.5 11.1086 0.0157489 *
## A:B:C 1 3.1 3.1 0.8571 0.3902585
## A:B:D 1 32.0 32.0 8.7771 0.0251971 *
## A:B:E 1 2.0 2.0 0.5486 0.4868642
## A:C:D 1 4.5 4.5 1.2343 0.3091018
## A:C:E 1 8.0 8.0 2.1943 0.1890296
## A:D:E 1 6.1 6.1 1.6800 0.2425450
## B:C:D 1 8.0 8.0 2.1943 0.1890296
## B:C:E 1 4.5 4.5 1.2343 0.3091018
## B:D:E 1 3.1 3.1 0.8571 0.3902585
## C:D:E 1 1.1 1.1 0.3086 0.5986433
## Residuals 6 21.9 3.6
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Ajuste do modelo reduzido com termos de 2 grau.
m2 <- update(m0, . ~ (A + B + C + D + E)^2)
anova(m2)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 1711.1 1711.1 290.0980 1.121e-11 ***
## B 1 10011.1 10011.1 1697.2503 < 2.2e-16 ***
## C 1 15.1 15.1 2.5642 0.1288617
## D 1 180.5 180.5 30.6013 4.553e-05 ***
## E 1 144.5 144.5 24.4980 0.0001449 ***
## A:B 1 1152.0 1152.0 195.3060 2.197e-10 ***
## A:C 1 12.5 12.5 2.1192 0.1648031
## A:D 1 3.1 3.1 0.5298 0.4772081
## A:E 1 3.1 3.1 0.5298 0.4772081
## B:C 1 8.0 8.0 1.3563 0.2612524
## B:D 1 0.1 0.1 0.0212 0.8860752
## B:E 1 0.1 0.1 0.0212 0.8860752
## C:D 1 10.1 10.1 1.7166 0.2086354
## C:E 1 3.1 3.1 0.5298 0.4772081
## D:E 1 40.5 40.5 6.8662 0.0185571 *
## Residuals 16 94.4 5.9
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Ajuste do modelo apenas com os termos relevantes.
m3 <- update(m0, . ~ A + B + D + E + A:B + D:E)
anova(m3)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 1711.1 1711.1 285.6636 3.436e-15 ***
## B 1 10011.1 10011.1 1671.3063 < 2.2e-16 ***
## D 1 180.5 180.5 30.1336 1.056e-05 ***
## E 1 144.5 144.5 24.1235 4.686e-05 ***
## A:B 1 1152.0 1152.0 192.3205 3.045e-13 ***
## D:E 1 40.5 40.5 6.7613 0.01542 *
## Residuals 25 149.8 6.0
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Razão entre modelos encaixados (apenas curiosidade).
anova(m3, m1)## Analysis of Variance Table
##
## Model 1: y ~ A + B + D + E + A:B + D:E
## Model 2: y ~ A + B + C + D + E + A:B + A:C + A:D + A:E + B:C + B:D + B:E +
## C:D + C:E + D:E + A:B:C + A:B:D + A:B:E + A:C:D + A:C:E +
## A:D:E + B:C:D + B:C:E + B:D:E + C:D:E
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 25 149.750
## 2 6 21.875 19 127.88 1.846 0.2299# Malha de valores para predição.
pred <- expand.grid(A = seq(-1, 1, by = 0.1),
B = seq(-1, 1, by = 0.1),
D = seq(-1, 1, by = 1),
E = seq(-1, 1, by = 1))
pred <- cbind(pred,
as.data.frame(predict(m3,
newdata = pred,
se.fit = TRUE)[1:2]))
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = A, y = B, z = fit, fill = fit)) +
facet_grid(facets = D ~ E) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black") +
coord_equal()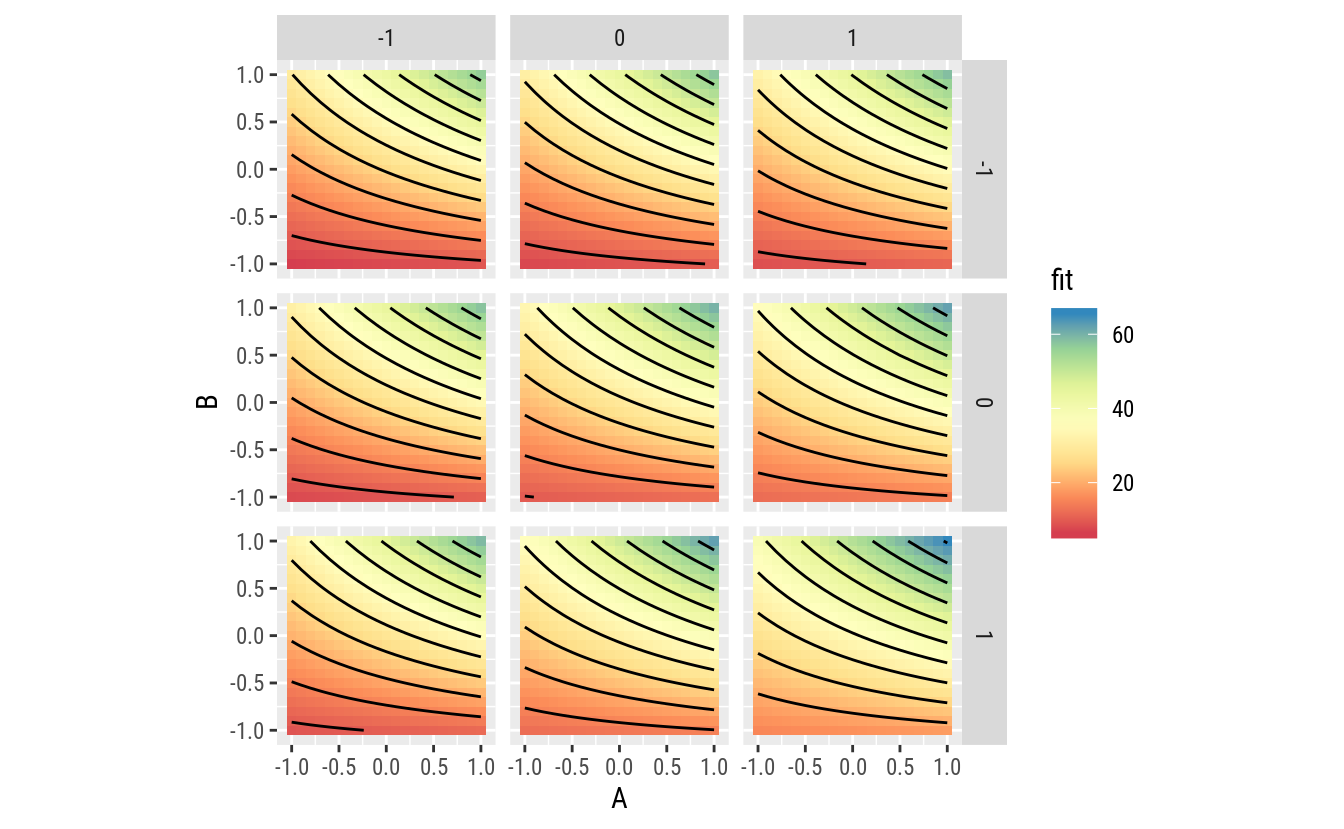
# Gráficos de linhas.
ggplot(data = pred,
mapping = aes(x = A, y = fit, color = B, group = B)) +
facet_grid(facets = D ~ E) +
geom_line() +
scale_color_distiller(palette = "BrBG")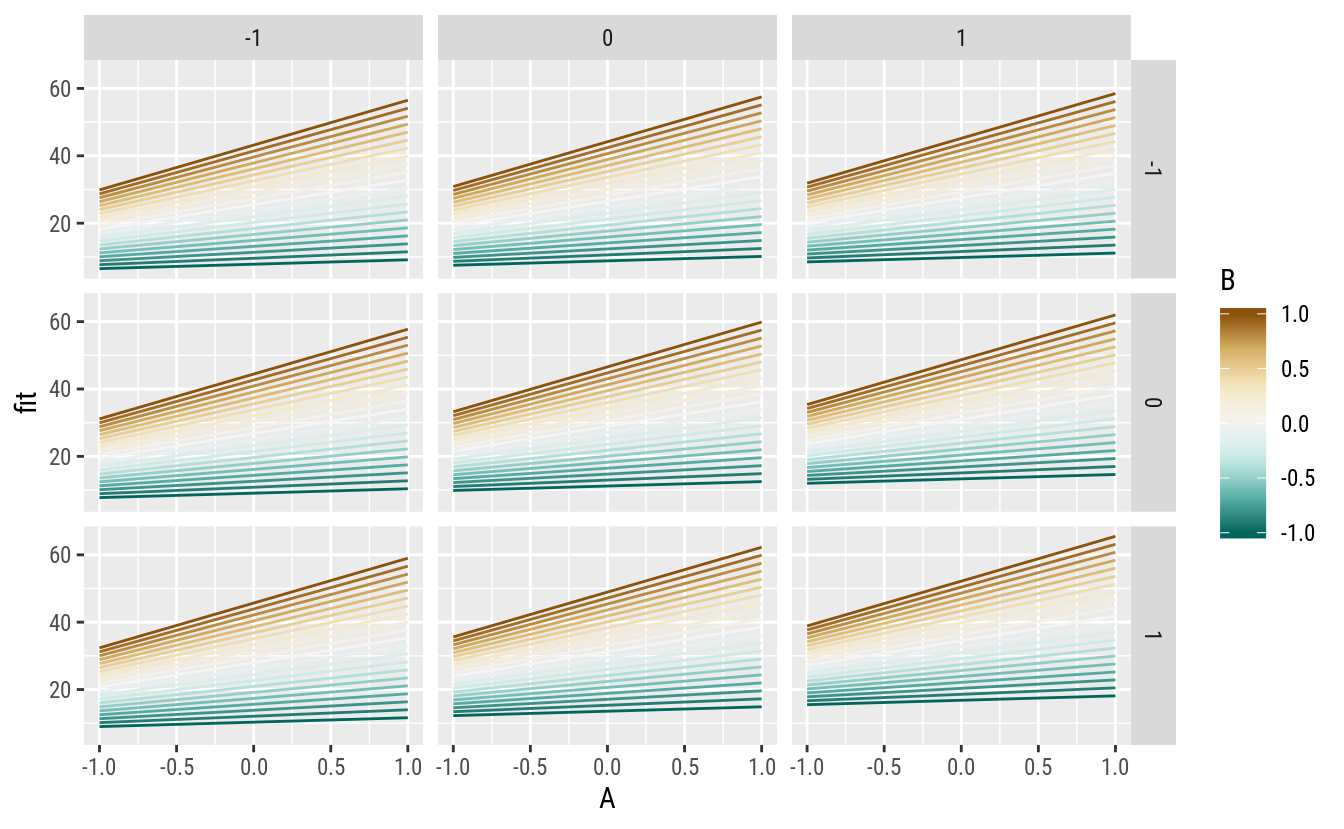
# Esquema de cores para o gráfico com a `lattice`.
colr <- RColorBrewer::brewer.pal(11, "Spectral")
colr <- colorRampPalette(colr, space = "rgb")
# Adicionando os contornos sobre a superfície. Requer funções externas.
source(paste0("https://raw.githubusercontent.com/walmes/wzRfun/",
"master/R/panel.3d.contour.R"))
# Gráfico de superfície em 3D com isolinhas (ainda não recomendo).
latticeExtra::useOuterStrips(
wireframe(fit ~ A + B | D + E,
data = pred,
as.table = TRUE,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100)))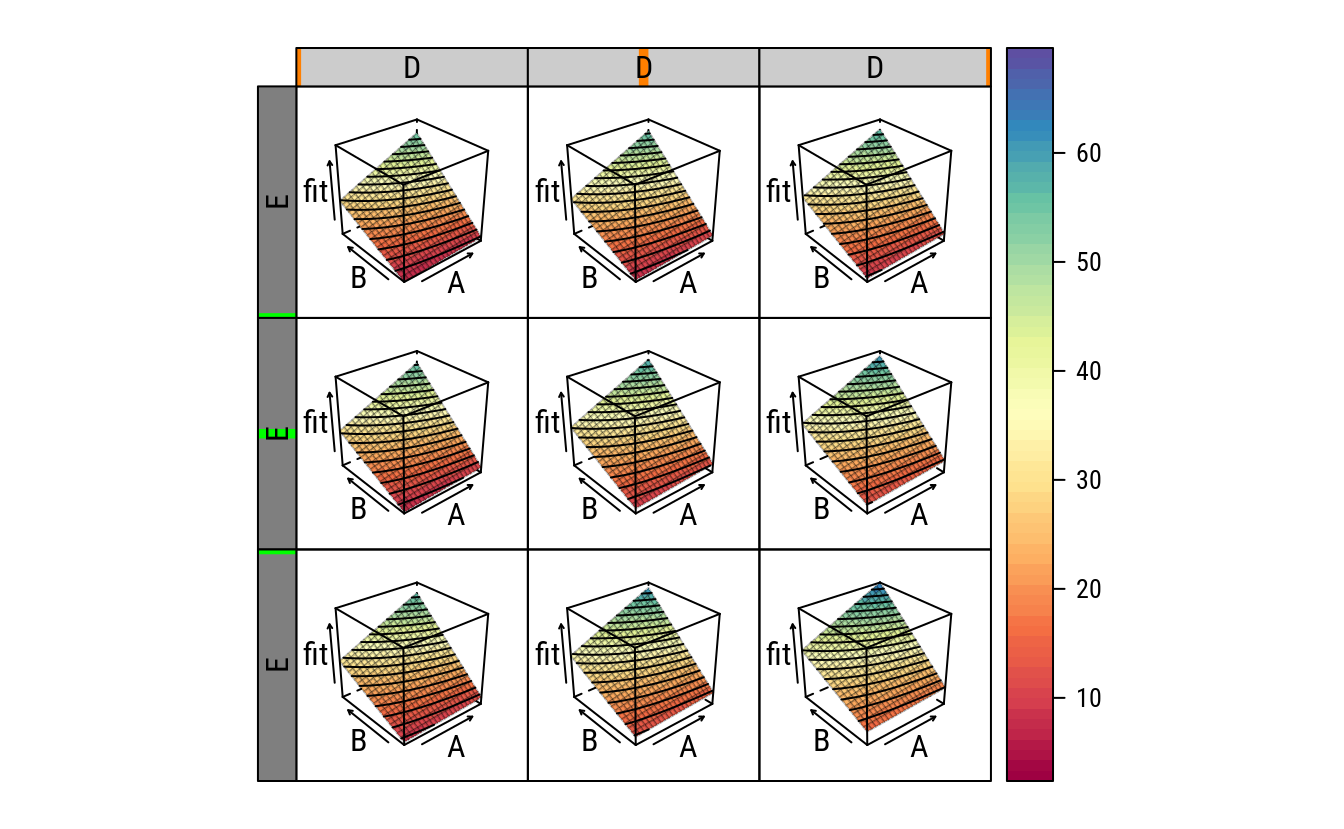
# Valores preditos em cada ponto experimental.
grid <- unique(ex14.14[, c("A", "B", "D", "E")])
grid$fit <- predict(m3, newdata = grid)
arrange(grid, fit)## A B D E fit
## 1 -1 -1 -1 -1 6.500
## 2 -1 -1 -1 1 8.500
## 3 -1 -1 1 -1 9.000
## 4 1 -1 -1 -1 9.125
## 5 1 -1 -1 1 11.125
## 6 1 -1 1 -1 11.625
## 7 -1 -1 1 1 15.500
## 8 1 -1 1 1 18.125
## 9 -1 1 -1 -1 29.875
## 10 -1 1 -1 1 31.875
## 11 -1 1 1 -1 32.375
## 12 -1 1 1 1 38.875
## 13 1 1 -1 -1 56.500
## 14 1 1 -1 1 58.500
## 15 1 1 1 -1 59.000
## 16 1 1 1 1 65.5004.2.3 Próximo experimento
Para obter a direção para um próximo experimento, será considerado o vetor gradiente no ponto \(\{1, 1, 1, 1\}\) nos fatores A, B, D e E. O modelo final tem a seguinte equação para os valores preditos \[ \hat{y} = f(x_1, x_2, x_4, x_5; \hat{\beta}) = \hat{\beta}_0 + \hat{\beta}_1 x_1 + \hat{\beta}_2 x_2 + \hat{\beta}_4 x_4 + \hat{\beta}_5 x_5 + \hat{\beta}_{12} x_1 x_2 + \hat{\beta}_{45} x_4 x_5. \]
Dessa forma, o vetor gradiente no ponto \(\{1, 1, 1, 1\}\) nos fatores A, B, D e E é \[ \begin{align*} \nabla f(x_1, x_2, x_4, x_5; \hat{\beta})\big |_{1, 1, 1, 1} &= \left( \frac{\mathrm{d} f}{\mathrm{d} x_1}, \frac{\mathrm{d} f}{\mathrm{d} x_2}, \frac{\mathrm{d} f}{\mathrm{d} x_4}, \frac{\mathrm{d} f}{\mathrm{d} x_5} \right)\bigg |_{1, 1, 1, 1}\\ &= (\hat{\beta}_1 + \hat{\beta}_{12}, \hat{\beta}_2 + \hat{\beta}_{12}, \hat{\beta}_4 + \hat{\beta}_{45}, \hat{\beta}_5 + \hat{\beta}_{45}). \end{align*} \]
O código abaixo determina o vetor gradiente na origem do delineamento e no ponto experimental abde. Como existe interação, o vetor gradiente muda com o ponto de suporte. Quando não há interação, o vetor gradiente é o mesmo em qualquer ponto.
# Estimativas dos parâmetros.
coef(m3)## (Intercept) A B D E
## 28.8750 7.3125 17.6875 2.3750 2.1250
## A:B D:E
## 6.0000 1.1250# Vetor gradiente em {0, 0, 0, 0}.
rbind(coef(m3)["A"],
coef(m3)["B"],
coef(m3)["D"],
coef(m3)["E"])## A
## [1,] 7.3125
## [2,] 17.6875
## [3,] 2.3750
## [4,] 2.1250# Vetor gradiente em {1, 1, 1, 1}.
rbind(coef(m3)["A"] + coef(m3)["A:B"],
coef(m3)["B"] + coef(m3)["A:B"],
coef(m3)["D"] + coef(m3)["D:E"],
coef(m3)["E"] + coef(m3)["D:E"])## A
## [1,] 13.3125
## [2,] 23.6875
## [3,] 3.5000
## [4,] 3.25004.3 Rendimento de uma reação
Esses dados são usados na seção 3.3 (página 129) do NETO et al. (2010). O experimento é um fatorial \(2^4\) para estudar o redimento (%) de uma reação em função dos fatores temperatura (A, 40 e 60 celsius), tipo de catalizador (B, I e II), concentração (C, 1 e 1.5 M) e pH (D, 7 e 6).
l <- c(-1, 1)
rend <- expand.grid(A = l, B = l, C = l, D = l,
KEEP.OUT.ATTRS = FALSE)
rend$y <- c(54, 85, 49, 62, 64, 94, 56, 70, 52, 87, 49, 64, 64, 94, 58,
73)4.3.1 Análise feita de forma operacional
Veja o código disponível na primeira seção. Apenas é necessário fazer a adaptação para o número de fatores.
4.3.2 Análise feita com funções
# Ajuste do modelo saturado (consome todos os graus de liberdade).
m0 <- lm(y ~ A * B * C * D, data = rend)
# NOTE: não é possível ver gráfico dos resíduos porque todos os resíduos
# são 0.
# # Quadro de análise de variância.
# anova(m0)
cfs <- coef(m0)[-1] %>%
enframe()
# Gráfico quantil-quantil normal das estimativas.
qq <- qqnorm(cfs$value, pch = 19, col = "orange")
text(x = qq$x, y = qq$y,
labels = cfs$name,
pos = ifelse(qq$x < 0, 4, 2))
ggplot(data = cfs,
mapping = aes(x = reorder(name, value), y = value)) +
geom_segment(aes(xend = name, yend = 0)) +
geom_segment(data = data.frame(x = 2.5, xend = 13.5, y = 2),
color = "orange",
arrow = arrow(length = unit(0.1, "inches"),
angle = 90,
ends = "both"),
mapping = aes(x = x, y = y, xend = xend, yend = y)) +
theme(axis.text.x = element_text(angle = 90, hjust = 1, vjust = 0.5))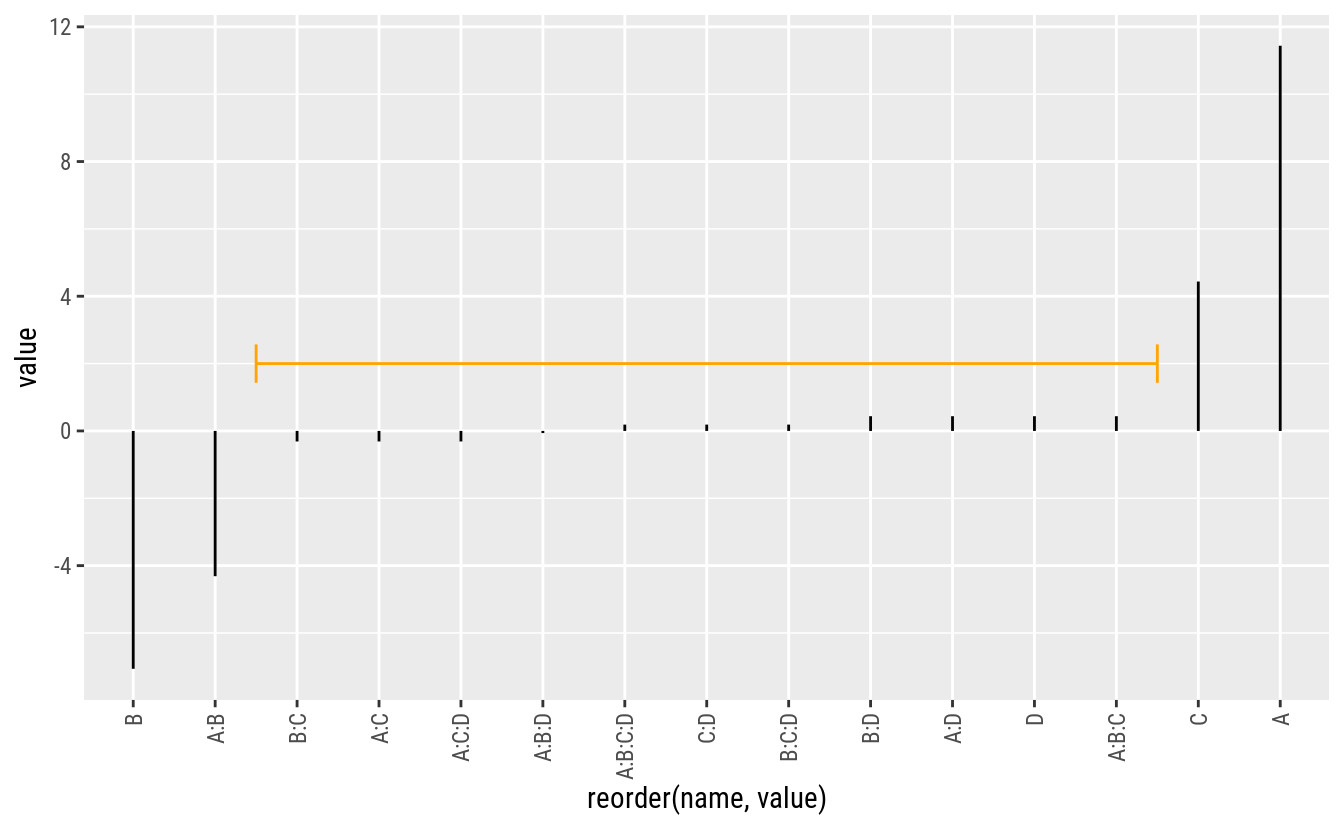
# Gráfico quantil-quantil normal nos efeitos estimados.
# FrF2::DanielPlot(m0, pch = 19)Pela análise do gráfico QQ-normal, os termos mais próximo de zero incluem as interações acima de 3 ordem. Pode-se então, para ter uma estimativa de variância residual, abandonar esses termos do modelo. Essa estimativa não é uma estimativa pura de variância residual mas sim inteiramente proveniente de termos omitidos do modelo.
# Ajuste do modelo reduzido com termos de 3 grau.
m1 <- update(m0, . ~ A + B + C + A:B)
anova(m1)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 2093.06 2093.06 1232.04 1.205e-12 ***
## B 1 798.06 798.06 469.76 2.253e-10 ***
## C 1 315.06 315.06 185.45 3.138e-08 ***
## A:B 1 297.56 297.56 175.15 4.226e-08 ***
## Residuals 11 18.69 1.70
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Malha de valores para predição.
pred <- expand.grid(A = seq(-1, 1, by = 0.1),
B = c(-1, 1),
C = seq(-1, 1, by = 0.1))
pred <- cbind(pred,
as.data.frame(predict(m1,
newdata = pred,
se.fit = TRUE)[1:2]))
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = A, y = C, z = fit, fill = fit)) +
facet_grid(facets = ~ B) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black") +
coord_equal()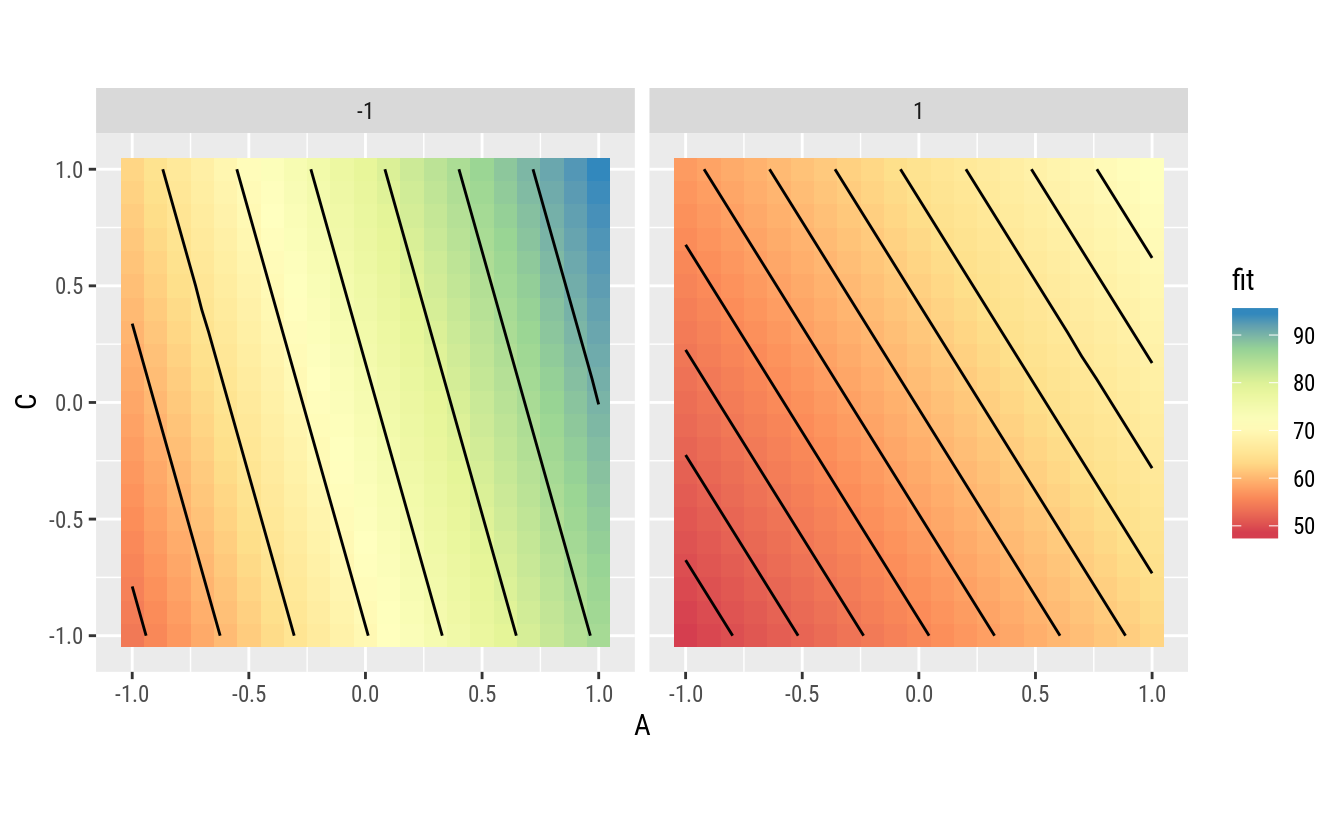
# Gráficos de linhas.
ggplot(data = pred,
mapping = aes(x = A, y = fit, color = C, group = C)) +
facet_grid(facets = ~ B) +
geom_line() +
scale_color_distiller(palette = "BrBG")
# Esquema de cores para o gráfico com a `lattice`.
colr <- RColorBrewer::brewer.pal(11, "Spectral")
colr <- colorRampPalette(colr, space = "rgb")
# Adicionando os contornos sobre a superfície. Requer funções externas.
source(paste0("https://raw.githubusercontent.com/walmes/wzRfun/",
"master/R/panel.3d.contour.R"))
# Gráfico de superfície em 3D com isolinhas (ainda não recomendo).
wireframe(fit ~ A + C | B,
data = pred,
as.table = TRUE,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100))
# Valores preditos em cada ponto experimental.
grid <- unique(rend[, c("A", "B", "C")])
grid$fit <- predict(m1, newdata = grid)
arrange(grid, fit)## A B C fit
## 1 -1 1 -1 48.5625
## 2 -1 -1 -1 54.0625
## 3 -1 1 1 57.4375
## 4 1 1 -1 62.8125
## 5 -1 -1 1 62.9375
## 6 1 1 1 71.6875
## 7 1 -1 -1 85.5625
## 8 1 -1 1 94.43754.3.3 Próximo experimento
Para obter a direção para um próximo experimento, será considerado o vetor gradiente no ponto \(\{1, 1\}\) nos fatores A e C considerando para B os dois tipos de catalizador, já que é um fator qualitativo. Por essa razão, não faz sentido um valor de B igual a 0 porque é uma variável qualitativa.
O modelo final tem a seguinte equação para os valores preditos \[ \hat{y} = f(x_1, x_2, x_3; \hat{\beta}) = \hat{\beta}_0 + \hat{\beta}_1 x_1 + \hat{\beta}_2 x_2 + \hat{\beta}_3 x_3 + \hat{\beta}_{12} x_1 x_2. \]
Dessa forma, o vetor gradiente no ponto \(\{1, 1\}\) nos fatores A, C, para os dois níveis de B é \[ \begin{align*} \nabla f(x_1, x_2, x_3; \hat{\beta}) &= \left( \frac{\mathrm{d} f}{\mathrm{d} x_1}, \frac{\mathrm{d} f}{\mathrm{d} x_2}, \frac{\mathrm{d} f}{\mathrm{d} x_3} \right)\\ \nabla f(x_1, x_2, x_3; \hat{\beta})\big |_{1, 1, 1} &= (\hat{\beta}_1 + \hat{\beta}_{12}, \hat{\beta}_2 + \hat{\beta}_{12}, \hat{\beta}_3)\\ \nabla f(x_1, x_2, x_3; \hat{\beta})\big |_{1, -1, 1} &= (\hat{\beta}_1 - \hat{\beta}_{12}, -\hat{\beta}_2 - \hat{\beta}_{12}, \hat{\beta}_3)\\ \end{align*} \]
O código abaixo determina o vetor gradiente nos pontos experimentais ac e abc do fatorial \(2^3\) em A, B e C . Como existe interação entre A e B, o vetor gradiente será diferente nestes dois pontos experimentais. No entanto, para um valor fixo de B, o vetor gradiente é o mesmo em qualquer ponto da superfície definida por A e C porque A e C não tem interação.
# Estimativas dos parâmetros.
coef(m1)## (Intercept) A B C A:B
## 67.1875 11.4375 -7.0625 4.4375 -4.3125# Vetor gradiente em A e C no ponto {1, -1, 1}.
gd_low <- rbind(coef(m1)["A"] - coef(m1)["A:B"],
coef(m1)["C"])
gd_low## A
## [1,] 15.7500
## [2,] 4.4375# Vetor gradiente em A e C no ponto {1, 1, 1}.
gd_hig <- rbind(coef(m1)["A"] + coef(m1)["A:B"],
coef(m1)["C"])
gd_hig## A
## [1,] 7.1250
## [2,] 4.4375# Malha de valores para predição.
pred <- expand.grid(A = seq(-1, 2, by = 0.1),
B = c(-1, 1),
C = seq(-1, 2, by = 0.1))
pred <- cbind(pred,
as.data.frame(predict(m1,
newdata = pred,
se.fit = TRUE)[1:2]))
# Quadrado para demarcar a região experimental.
design_square <- data.frame(x0 = -1, x1 = 1, y0 = -1, y1 = 1)
rect_design <- geom_rect(data = design_square,
inherit.aes = FALSE,
mapping = aes(xmin = x0, ymin = y0,
xmax = x1, ymax = y1),
color = "black",
fill = NA,
linetype = 2)
# Gradientes par cada nível de B.
grad <- data.frame(B = c(-1, 1),
x = 0,
xend = c(gd_low[1], gd_hig[1])/10,
y = 0,
yend = c(gd_low[2], gd_hig[2])/10)
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = A, y = C, z = fit, fill = fit)) +
facet_grid(facets = ~ B) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black", size = 0.2) +
rect_design +
geom_segment(data = grad,
color = "purple",
arrow = arrow(length = unit(0.1, "inches"),
angle = 30,
ends = "last"),
mapping = aes(x = x,
y = y,
xend = xend,
yend = yend,
z = NULL, fill = NULL)) +
coord_equal()
4.4 Separação de gases por adsorção
No desenvolvimento em laboratório de um processo de enriquecimento de gases por adsorção, usou-se um planejamento \(2^4\) para investigar a influência de quatro fatores na produtividade do adsorvente (mol kg-1 ciclo-1).
| Fator | Nível baixo | Nível alto |
|---|---|---|
| Pressão de adsorção (bar) | 1,40 | 2,40 |
| Pressão de dessorção (bar) | 0,05 | 0,20 |
| Vazão de alimentação (m3 h-1) | 0,10 | 0,30 |
| Tempo de adsorção (s) | 8 | 30 |
# Criação dos dados.
l <- c(-1, 1)
tb3a <- expand.grid(A = l, B = l, C = l, D = l,
KEEP.OUT.ATTRS = FALSE)
tb3a$y <- c(275, 315, 287, 355, 465, 585, 540, 630, 595, 655, 560, 675,
1150, 1300, 1250, 1400)/1004.4.1 Análise feita com funções
# Ajuste do modelo saturado (consome todos os graus de liberdade).
m0 <- lm(y ~ A * B * C * D, data = tb3a)
# NOTE: não é possível ver gráfico dos resíduos porque todos os resíduos
# são 0.
# # Quadro de análise de variância.
# anova(m0)
cfs <- coef(m0)[-1] %>%
enframe()
# Gráfico quantil-quantil normal das estimativas.
qq <- qqnorm(cfs$value, pch = 19, col = "orange")
text(x = qq$x, y = qq$y,
labels = cfs$name,
pos = ifelse(qq$x < 0, 4, 2))
ggplot(data = cfs,
mapping = aes(x = reorder(name, value), y = value)) +
geom_segment(aes(xend = name, yend = 0)) +
geom_segment(data = data.frame(x = 1.5, xend = 10.5, y = 0.5),
color = "orange",
arrow = arrow(length = unit(0.1, "inches"),
angle = 90,
ends = "both"),
mapping = aes(x = x, y = y, xend = xend, yend = y)) +
theme(axis.text.x = element_text(angle = 90, hjust = 1, vjust = 0.5))
# Gráfico quantil-quantil normal nos efeitos estimados.
# FrF2::DanielPlot(m0, pch = 19)Pela análise do gráfico QQ-normal, os termos mais próximo de zero incluem as interações acima de 3 ordem. Pode-se então, para ter uma estimativa de variância residual, abandonar esses termos do modelo. Essa estimativa não é uma estimativa pura de variância residual mas sim inteiramente proveniente de termos omitidos do modelo.
# Ajuste do modelo reduzido com termos de 2 grau.
m1 <- update(m0, . ~ (A + B + C + D)^2)
anova(m1)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 3.930 3.930 82.3856 0.0002715 ***
## B 1 0.797 0.797 16.6971 0.0094820 **
## C 1 81.135 81.135 1700.7217 1.581e-07 ***
## D 1 106.761 106.761 2237.8736 7.973e-08 ***
## A:B 1 0.018 0.018 0.3680 0.5705797
## A:C 1 0.322 0.322 6.7508 0.0483526 *
## A:D 1 0.154 0.154 3.2293 0.1322647
## B:C 1 0.501 0.501 10.4925 0.0229695 *
## B:D 1 0.001 0.001 0.0221 0.8875285
## C:D 1 16.545 16.545 346.8006 8.218e-06 ***
## Residuals 5 0.239 0.048
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Refina mais o modelo.
m2 <- update(m0, . ~ A + B + C + D + B:C + C:D)
anova(m2)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 3.930 3.930 48.2406 6.718e-05 ***
## B 1 0.797 0.797 9.7769 0.01218 *
## C 1 81.135 81.135 995.8531 1.581e-10 ***
## D 1 106.761 106.761 1310.3809 4.639e-11 ***
## B:C 1 0.501 0.501 6.1438 0.03507 *
## C:D 1 16.545 16.545 203.0682 1.760e-07 ***
## Residuals 9 0.733 0.081
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Malha de valores para predição.
pred <- expand.grid(A = c(-1, 1),
B = c(-1, 1),
C = seq(-1, 1, by = 0.1),
D = seq(-1, 1, by = 0.1))
pred <- cbind(pred,
as.data.frame(predict(m2,
newdata = pred,
se.fit = TRUE)[1:2]))
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = C, y = D, z = fit, fill = fit)) +
facet_grid(facets = A ~ B) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black") +
coord_equal()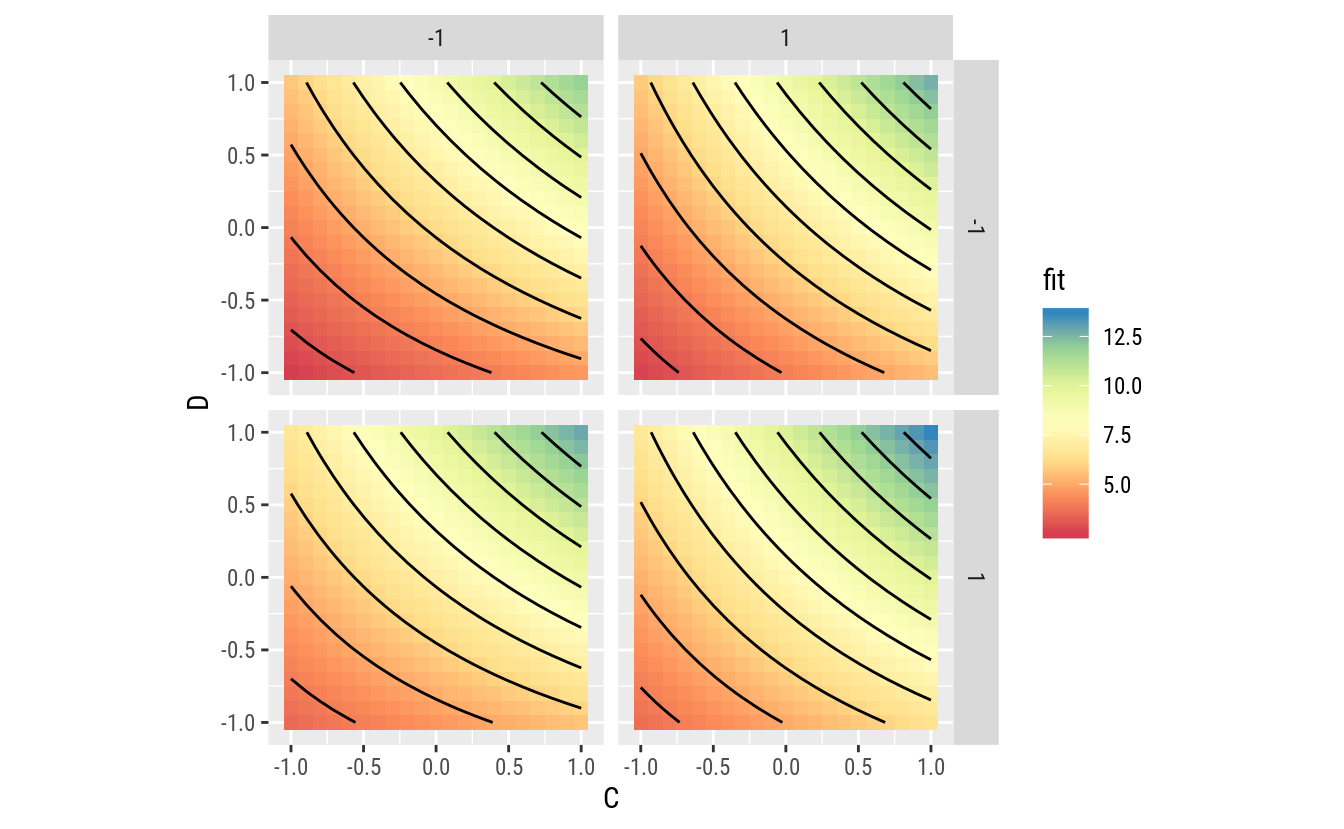
# Gráficos de linhas.
ggplot(data = pred,
mapping = aes(x = C, y = fit, color = D, group = D)) +
facet_grid(facets = A ~ B) +
geom_line() +
scale_color_distiller(palette = "BrBG")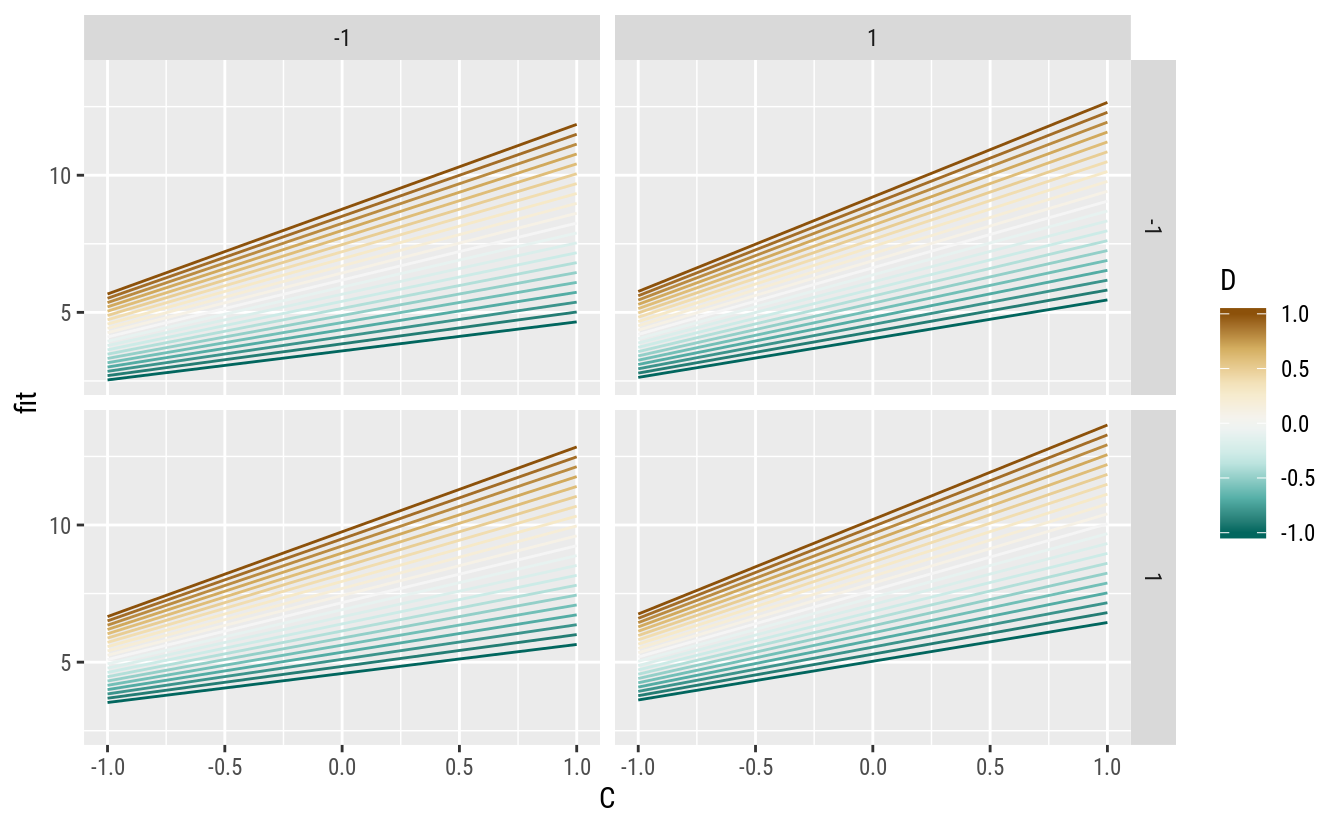
# Esquema de cores para o gráfico com a `lattice`.
colr <- RColorBrewer::brewer.pal(11, "Spectral")
colr <- colorRampPalette(colr, space = "rgb")
# Adicionando os contornos sobre a superfície. Requer funções externas.
source(paste0("https://raw.githubusercontent.com/walmes/wzRfun/",
"master/R/panel.3d.contour.R"))
# Gráfico de superfície em 3D com isolinhas (ainda não recomendo).
latticeExtra::useOuterStrips(
wireframe(fit ~ C + D| A + B,
data = pred,
as.table = TRUE,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100)))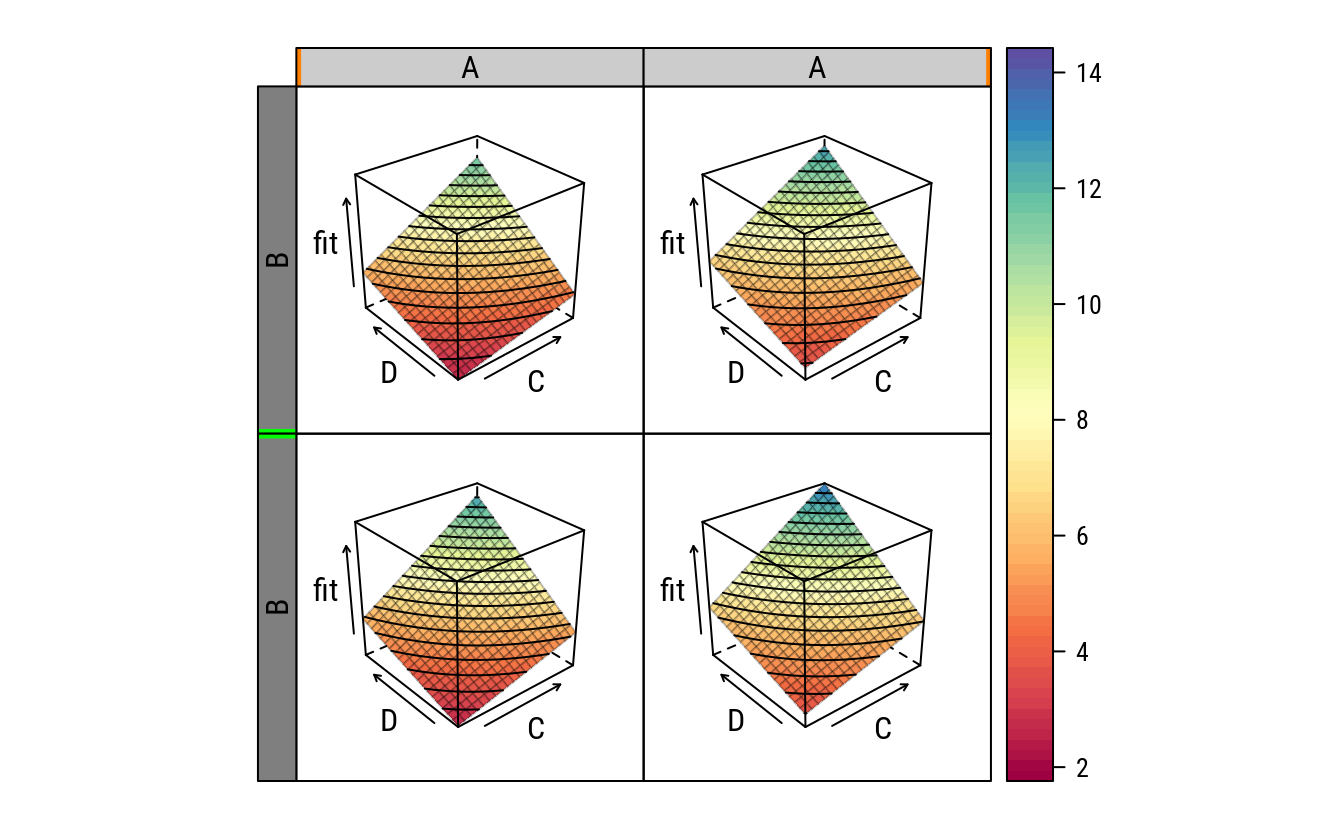
# Valores preditos em cada ponto experimental.
grid <- unique(tb3a[, c("A", "B", "C", "D")])
grid$fit <- predict(m2, newdata = grid)
arrange(grid, fit)## A B C D fit
## 1 -1 -1 -1 -1 2.538125
## 2 -1 1 -1 -1 2.630625
## 3 1 -1 -1 -1 3.529375
## 4 1 1 -1 -1 3.621875
## 5 -1 -1 1 -1 4.654375
## 6 -1 1 1 -1 5.454375
## 7 1 -1 1 -1 5.645625
## 8 -1 -1 -1 1 5.670625
## 9 -1 1 -1 1 5.763125
## 10 1 1 1 -1 6.445625
## 11 1 -1 -1 1 6.661875
## 12 1 1 -1 1 6.754375
## 13 -1 -1 1 1 11.854375
## 14 -1 1 1 1 12.654375
## 15 1 -1 1 1 12.845625
## 16 1 1 1 1 13.6456254.4.2 Próximo experimento
Para obter a direção para um próximo experimento, será considerado o vetor gradiente no ponto \(\{0, 0, 0, 0\}\) em todos os fatores.
O modelo final tem a seguinte equação para os valores preditos \[ \hat{y} = f(x_1, x_2, x_3, x_4; \hat{\beta}) = \hat{\beta}_0 + \hat{\beta}_1 x_1 + \hat{\beta}_2 x_2 + \hat{\beta}_3 x_3 + \hat{\beta}_4 x_4 + \hat{\beta}_{23} x_2 x_3 + \hat{\beta}_{34} x_3 x_4. \]
Dessa forma, o vetor gradiente no ponto central é simplesmente as estimativas dos parâmetros que descrevem os efeitos principais \[ \begin{align*} \nabla f(x_1, x_2, x_3, x_4; \hat{\beta}) &= \left( \frac{\mathrm{d} f}{\mathrm{d} x_1}, \frac{\mathrm{d} f}{\mathrm{d} x_2}, \frac{\mathrm{d} f}{\mathrm{d} x_3}, \frac{\mathrm{d} f}{\mathrm{d} x_4} \right)\\ \nabla f(x_1, x_2, x_3, x_4; \hat{\beta})\big |_{0, 0, 0, 0} &= (\hat{\beta}_1, \hat{\beta}_2, \hat{\beta}_3, \hat{\beta}_4). \end{align*} \]
O fragmento abaixo calcula pontos na direção indicada pelo vetor gradiente. Novos experimentos podem ser feitos tendo esses valores como coordenadas do centro do experimento. Cabe ao experimentador decidir qual será o centro do experimento nessa direção e se o experimento irá manter a amplitude entre os níveis do fatores. O experimentador pode aumentar a diferença entre os níveis para avaliar uma região experimental mais ampla.
# Vetor gradiente.
coef(m1)[names(tb3a)[1:4]]## A B C D
## 0.495625 0.223125 2.251875 2.583125# Valores que podem ser o centro de novos experimentos.
delta <- seq(0, 2, by = 0.25)
# Centro para novos experimentos.
new_des <- outer(delta,
coef(m1)[names(tb3a)[1:4]],
FUN = "*")
new_des## A B C D
## [1,] 0.0000000 0.00000000 0.0000000 0.0000000
## [2,] 0.1239063 0.05578125 0.5629687 0.6457812
## [3,] 0.2478125 0.11156250 1.1259375 1.2915625
## [4,] 0.3717188 0.16734375 1.6889062 1.9373437
## [5,] 0.4956250 0.22312500 2.2518750 2.5831250
## [6,] 0.6195313 0.27890625 2.8148437 3.2289062
## [7,] 0.7434375 0.33468750 3.3778125 3.8746875
## [8,] 0.8673438 0.39046875 3.9407812 4.5204688
## [9,] 0.9912500 0.44625000 4.5037500 5.1662500# Para passar da escala codificada para escala real.
decode <- function(z, xmin, xmax) {
r <- 0.5 * (xmax - xmin)
m <- 0.5 * (xmax + xmin)
z * r + m
}
# Centro de novos experimentos na escala original.
cbind(A = decode(new_des[, "A"], 1.40, 2.40),
B = decode(new_des[, "B"], 0.05, 0.20),
C = decode(new_des[, "C"], 0.10, 0.30),
D = decode(new_des[, "D"], 8, 30))## A B C D
## [1,] 1.900000 0.1250000 0.2000000 19.00000
## [2,] 1.961953 0.1291836 0.2562969 26.10359
## [3,] 2.023906 0.1333672 0.3125938 33.20719
## [4,] 2.085859 0.1375508 0.3688906 40.31078
## [5,] 2.147813 0.1417344 0.4251875 47.41437
## [6,] 2.209766 0.1459180 0.4814844 54.51797
## [7,] 2.271719 0.1501016 0.5377812 61.62156
## [8,] 2.333672 0.1542852 0.5940781 68.72516
## [9,] 2.395625 0.1584688 0.6503750 75.82875Referências Bibliográficas
MONTGOMERY, D. C.; RUNGER, G. C. Estatística aplicada e probabilidade para engenheiros. Rio de Janeiro: Livros Técnicos e Científicos, 2009.
NETO, B.; SCARMINIO, I.; BRUNS, R. Como fazer experimentos: Pesquisa e desenvolvimento na ciência e na indústria. 4th ed. Bookman, 2010.