Capítulo 3 Análise do fatorial 2k com repetições
library(tidyverse)
library(lattice)3.1 Tempo de vida de ferramenta de corte
Um engenheiro está interessado no efeito da velocidade de corte (A), na dureza do metal (B) e no ângulo de corte (C) sobre a vida de uma ferramenta de corte (y). Dois níveis de cada fator são escolhidos e duas réplicas de um planejamento fatorial \(2^3\) são feitas. Os dados de vida (em horas), da ferramenta são recriados nos fragmentos de código abaixo.
Esses dados são do exerício 14-12, na página 353 do MONTGOMERY; RUNGER (2009). Em MONTGOMERY (2001), dados do mesmo experimento são reportados com 3 repetições na página 276, exercício 6-1, mas a resposta está dividida por 10.
3.1.1 Análise exploratória
Nessa seção serão feitos os cálculos para obter as estimativas dos parâmetros, somas de quadrados, predição e erros padrões. Na seção seguinte os dados serão analisados com funções do R e será feita discussão dos resultados.
rm(list = objects())
# Níveis codificados.
l <- c(-1, 1)
# Tabela contendo todos os pontos experimentais e repetições.
ex14.12 <- expand.grid(A = l,
B = l,
C = l,
rept = 1:2,
KEEP.OUT.ATTRS = FALSE)
# Variável resposta do experimento.
# ex41.12$y <- scan()
# dput(ex41.12$y)
ex14.12$y <- c(221, 325, 354, 552, 440, 406, 605, 392, 311, 435, 348,
472, 453, 377, 500, 419)
# Estrutura.
str(ex14.12)## 'data.frame': 16 obs. of 5 variables:
## $ A : num -1 1 -1 1 -1 1 -1 1 -1 1 ...
## $ B : num -1 -1 1 1 -1 -1 1 1 -1 -1 ...
## $ C : num -1 -1 -1 -1 1 1 1 1 -1 -1 ...
## $ rept: int 1 1 1 1 1 1 1 1 2 2 ...
## $ y : num 221 325 354 552 440 406 605 392 311 435 ...# Repetições de cada ponto experimental.
ftable(xtabs(~A + B + C, data = ex14.12))## C -1 1
## A B
## -1 -1 2 2
## 1 2 2
## 1 -1 2 2
## 1 2 2gg1 <-
ggplot(data = ex14.12,
mapping = aes(x = A, y = y, color = B)) +
facet_wrap(facets = ~C) +
geom_point() +
stat_summary(mapping = aes(group = B),
fun.y = "mean",
geom = "line")
gg2 <-
ggplot(data = ex14.12,
mapping = aes(x = C, y = y, color = B)) +
facet_wrap(facets = ~A) +
geom_point() +
stat_summary(mapping = aes(group = B),
fun.y = "mean",
geom = "line")
gridExtra::grid.arrange(gg1, gg2, ncol = 1)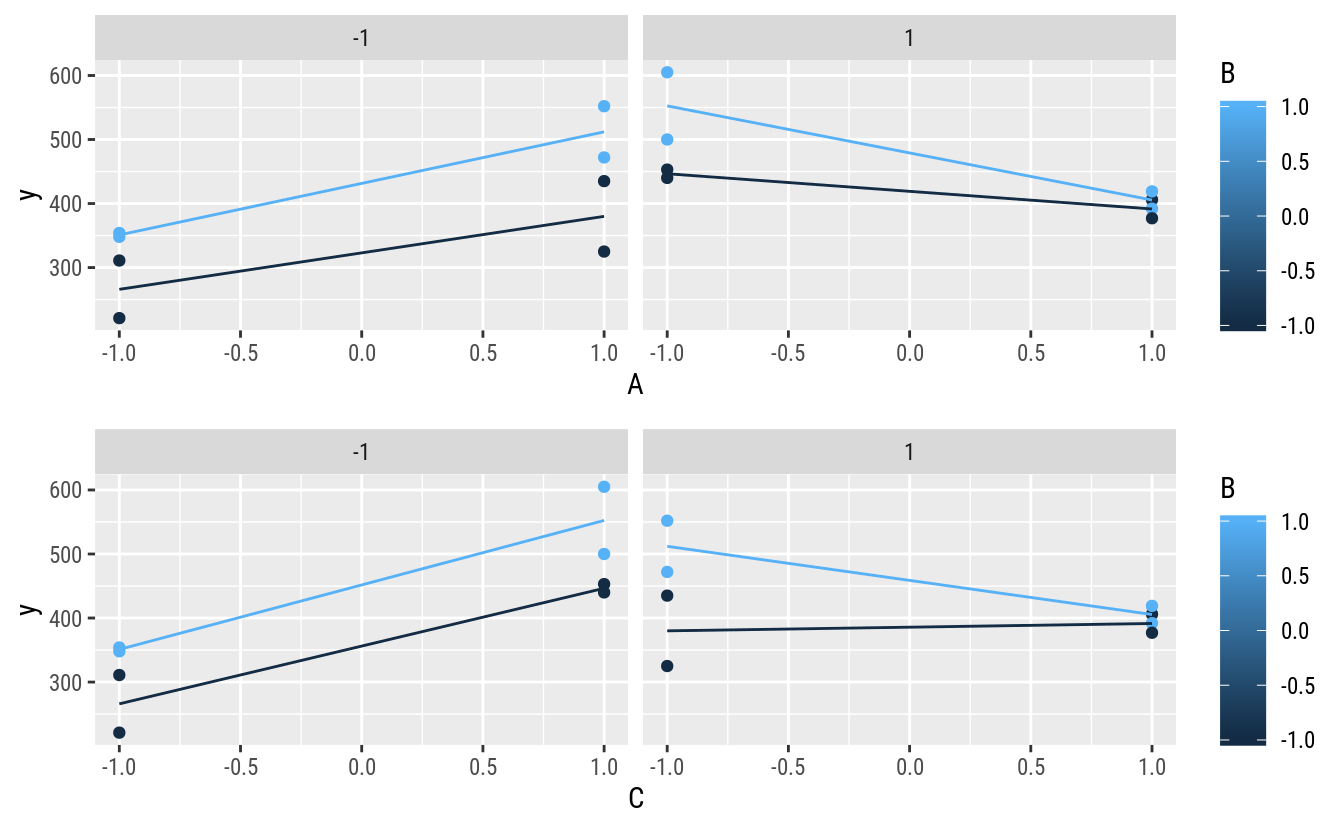
3.1.2 Análise feita de forma operacional
# Tabela de dados.
ex14.12## A B C rept y
## 1 -1 -1 -1 1 221
## 2 1 -1 -1 1 325
## 3 -1 1 -1 1 354
## 4 1 1 -1 1 552
## 5 -1 -1 1 1 440
## 6 1 -1 1 1 406
## 7 -1 1 1 1 605
## 8 1 1 1 1 392
## 9 -1 -1 -1 2 311
## 10 1 -1 -1 2 435
## 11 -1 1 -1 2 348
## 12 1 1 -1 2 472
## 13 -1 -1 1 2 453
## 14 1 -1 1 2 377
## 15 -1 1 1 2 500
## 16 1 1 1 2 419# Matriz do modelo e vetor resposta.
# X <- model.matrix(~A * B * C, data = ex14.12)
X <- with(ex14.12,
cbind(I = 1,
A, B, C,
AB = A * B, AC = A * C, BC = B * C,
ABC = A * B * C))
y <- cbind(ex14.12$y)
# Matriz do modelo ao lado do vetor da resposta.
data.frame(X, "|" = "|", y, check.names = FALSE)## I A B C AB AC BC ABC | y
## 1 1 -1 -1 -1 1 1 1 -1 | 221
## 2 1 1 -1 -1 -1 -1 1 1 | 325
## 3 1 -1 1 -1 -1 1 -1 1 | 354
## 4 1 1 1 -1 1 -1 -1 -1 | 552
## 5 1 -1 -1 1 1 -1 -1 1 | 440
## 6 1 1 -1 1 -1 1 -1 -1 | 406
## 7 1 -1 1 1 -1 -1 1 -1 | 605
## 8 1 1 1 1 1 1 1 1 | 392
## 9 1 -1 -1 -1 1 1 1 -1 | 311
## 10 1 1 -1 -1 -1 -1 1 1 | 435
## 11 1 -1 1 -1 -1 1 -1 1 | 348
## 12 1 1 1 -1 1 -1 -1 -1 | 472
## 13 1 -1 -1 1 1 -1 -1 1 | 453
## 14 1 1 -1 1 -1 1 -1 -1 | 377
## 15 1 -1 1 1 -1 -1 1 -1 | 500
## 16 1 1 1 1 1 1 1 1 | 419k <- 3 # Número de fatores.
r <- 2 # Número de repetições.
# Contrates são o principal artefato. É apenas X'y.
ctr <- crossprod(X, y)
ctr## [,1]
## I 6610
## A 146
## B 674
## C 574
## AB -90
## AC -954
## BC -194
## ABC -278# Estimativas dos parâmetros.
# beta <- solve(crossprod(X), crossprod(X, y))
beta <- diag(1/(r * 2^k), 2^k) %*% ctr
beta## [,1]
## [1,] 413.125
## [2,] 9.125
## [3,] 42.125
## [4,] 35.875
## [5,] -5.625
## [6,] -59.625
## [7,] -12.125
## [8,] -17.375# Estimativa dos parâmetros (por somatórios basicamente).
ctr/(r * 2^k) # idem ao `beta`## [,1]
## I 413.125
## A 9.125
## B 42.125
## C 35.875
## AB -5.625
## AC -59.625
## BC -12.125
## ABC -17.375# Somas de quadrados.
tail(ctr, n = -1)^2/(r * 2^k)## [,1]
## A 1332.25
## B 28392.25
## C 20592.25
## AB 506.25
## AC 56882.25
## BC 2352.25
## ABC 4830.25# Valores ajustados para cada ponto experimental.
ex14.12$hy <- X %*% beta
# Resíduos.
ex14.12$res <- with(ex14.12, y - hy)
# Desvio padrão residual.
s2 <- with(ex14.12, sum(res^2)/(nrow(X) - ncol(X)))
s2## [1] 2462.5# Erros padrões das estimativas dos parâmetros.
# summary(m0)$coefficients
sqrt(s2/(r * 2^k))## [1] 12.4059# Erros padrões das médias nos pontos experimentais.
# predict(m0, se.fit = TRUE)$se.fit
sqrt(s2/r)## [1] 35.08917# Matriz para a predição de um ponto qualquer.
# Xnew <- model.matrix(~A * B * C,
# data = data.frame(A = -1, B = 0.25, C = 0.5))
Xnew <- with(data.frame(A = -1, B = 0.25, C = 0.5),
cbind(I = 1,
A, B, C,
AB = A * B, AC = A * C, BC = B * C,
ABC = A * B * C))
Xnew## I A B C AB AC BC ABC
## [1,] 1 -1 0.25 0.5 -0.25 -0.5 0.125 -0.125# Predição em um ponto qualquer.
Xnew %*% beta## [,1]
## [1,] 464.3438# Variância da predição em um ponto qualquer.
# Xnew %*% (s2 * solve(crossprod(X))) %*% t(Xnew)
Xnew %*% (s2 * diag(1/(r * 2^k), 2^k)) %*% t(Xnew)## [,1]
## [1,] 408.81353.1.3 Análise feita com funções
Nessa sessão serão usadas as funções do R para análise dos dados. Como é um modelo linear, usa-se a lm().
# Ajuste do modelo saturado.
m0 <- lm(y ~ A * B * C, data = ex14.12)
# Diagnóstico nos resíduos.
par(mfrow = c(1, 2))
plot(m0, which = 3)
plot(m0, which = 2)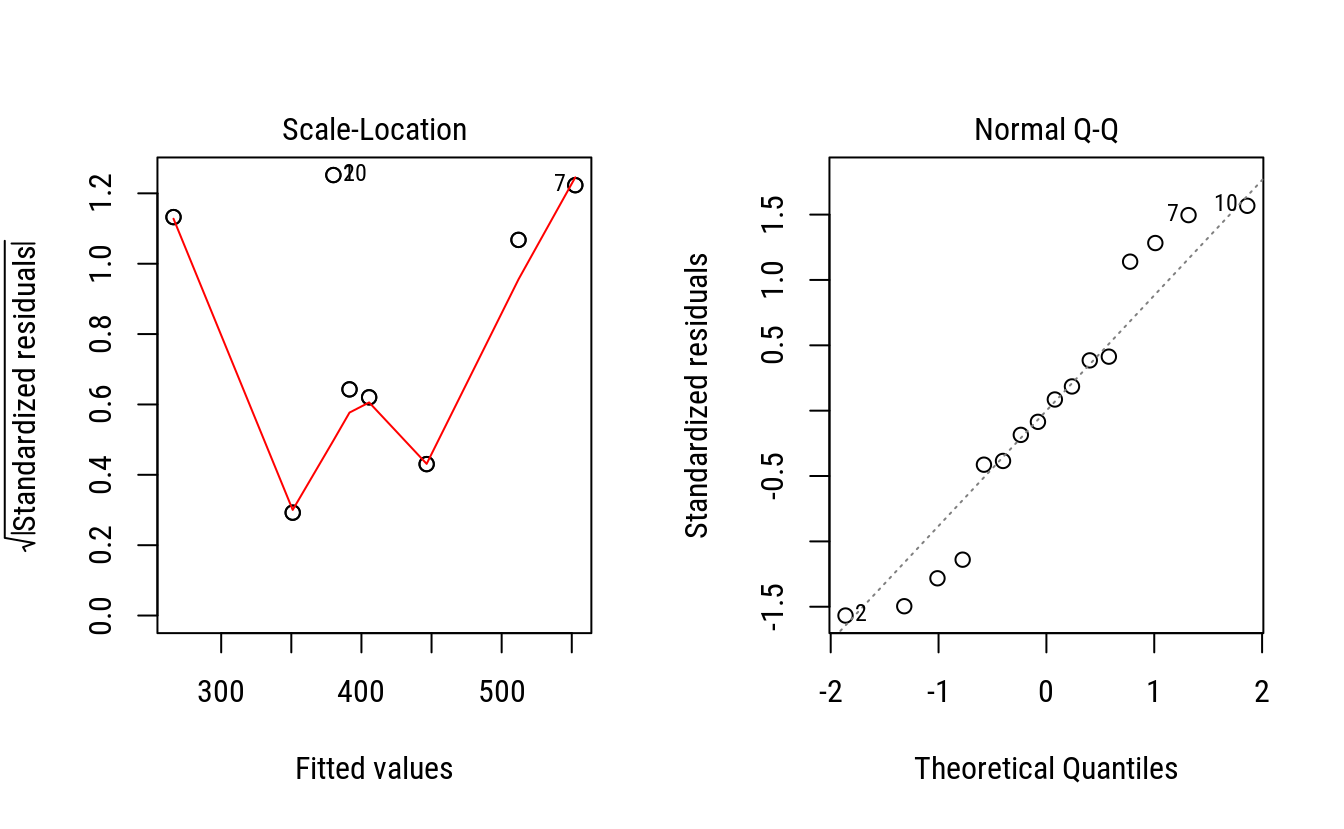
layout(1)Pela inspeção dos gráficos, não existem evidências contra o atendimento dos pressupostos. Obviamente que por serem poucas observações, dificulta-se detectar padrões característicos, como relação média-variância.
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 1332 1332 0.5410 0.483017
## B 1 28392 28392 11.5298 0.009422 **
## C 1 20592 20592 8.3623 0.020144 *
## A:B 1 506 506 0.2056 0.662297
## A:C 1 56882 56882 23.0994 0.001345 **
## B:C 1 2352 2352 0.9552 0.357017
## A:B:C 1 4830 4830 1.9615 0.198923
## Residuals 8 19700 2463
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas.
summary(m0)##
## Call:
## lm(formula = y ~ A * B * C, data = ex14.12)
##
## Residuals:
## Min 1Q Median 3Q Max
## -55.00 -20.88 0.00 20.88 55.00
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 413.125 12.406 33.301 7.22e-10 ***
## A 9.125 12.406 0.736 0.48302
## B 42.125 12.406 3.396 0.00942 **
## C 35.875 12.406 2.892 0.02014 *
## A:B -5.625 12.406 -0.453 0.66230
## A:C -59.625 12.406 -4.806 0.00134 **
## B:C -12.125 12.406 -0.977 0.35702
## A:B:C -17.375 12.406 -1.401 0.19892
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 49.62 on 8 degrees of freedom
## Multiple R-squared: 0.8536, Adjusted R-squared: 0.7256
## F-statistic: 6.665 on 7 and 8 DF, p-value: 0.007899Pela avaliação do quadro de análise de variância, não houve interações relevantes envolvendo B, apenas seu efeito principal. Houve interação dupla entre A e C. Portanto, alguns termos do modelo podem ser abandonados.
Em termos de hipóteses, para delineamentos de efeitos ortogonais e fatores de dois níveis, a estatística F da anova() e t do summary(), são essencialmente a mesma coisa.
# Ajuste do modelo reduzido apenas nos termos relevantes.
m1 <- update(m0, . ~ B + A * C)
anova(m1)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## B 1 28392 28392 11.4030 0.0061770 **
## A 1 1332 1332 0.5351 0.4797786
## C 1 20592 20592 8.2704 0.0150836 *
## A:C 1 56882 56882 22.8453 0.0005717 ***
## Residuals 11 27389 2490
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Variâncias residuais.
c(s2_m0 = summary(m0)$sigma^2,
s2_m1 = summary(m1)$sigma^2)## s2_m0 s2_m1
## 2462.500 2489.886# Teste para nulidade dos termos abandonados.
anova(m1, m0)## Analysis of Variance Table
##
## Model 1: y ~ B + A + C + A:C
## Model 2: y ~ A * B * C
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 11 27389
## 2 8 19700 3 7688.8 1.0408 0.4254O modelo reduzido ajustado pelo abandono daqueles não relevantes não diferiu, conforme esperado, do modelo maximal. O negligenciável aumento da variância resídual não é nenhum problema. Poderia-se, sem problema algum, conduzir as inferência com a estimativa pura da variância, proveniente do modelo m0. Por outro lado, não haveriam diferenças substanciais nos resultados.
# Malha fina de valores para predição.
pred <- expand.grid(A = seq(-1, 1, by = 0.1),
C = seq(-1, 1, by = 0.1),
B = seq(-1, 1, by = 0.5),
KEEP.OUT.ATTRS = FALSE)
pred <- cbind(pred,
as.data.frame(predict(m1,
newdata = pred,
se.fit = TRUE)[1:2]))
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = A, y = C, z = fit, fill = fit)) +
facet_wrap(facets = ~B) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black") +
coord_equal()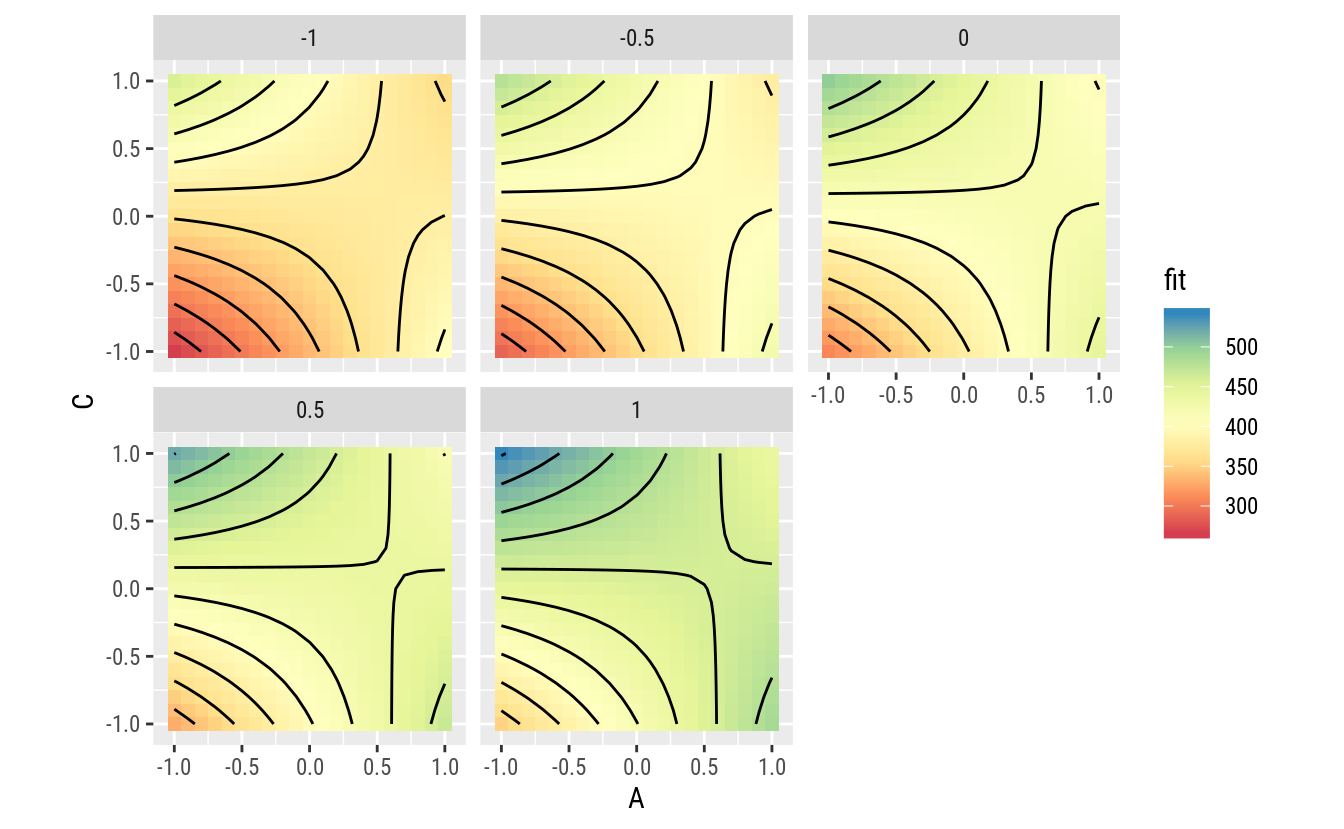
A função apresenta um comportamento que, conforme antecipado, indica que o efeito de A depende do valor de C e vice versa. É possível entender melhor com gráfico de linhas.
# Gráficos de linhas.
ggplot(data = pred,
mapping = aes(x = A, y = fit, color = C, group = C)) +
facet_wrap(facets = ~B) +
geom_line() +
scale_color_distiller(palette = "PRGn")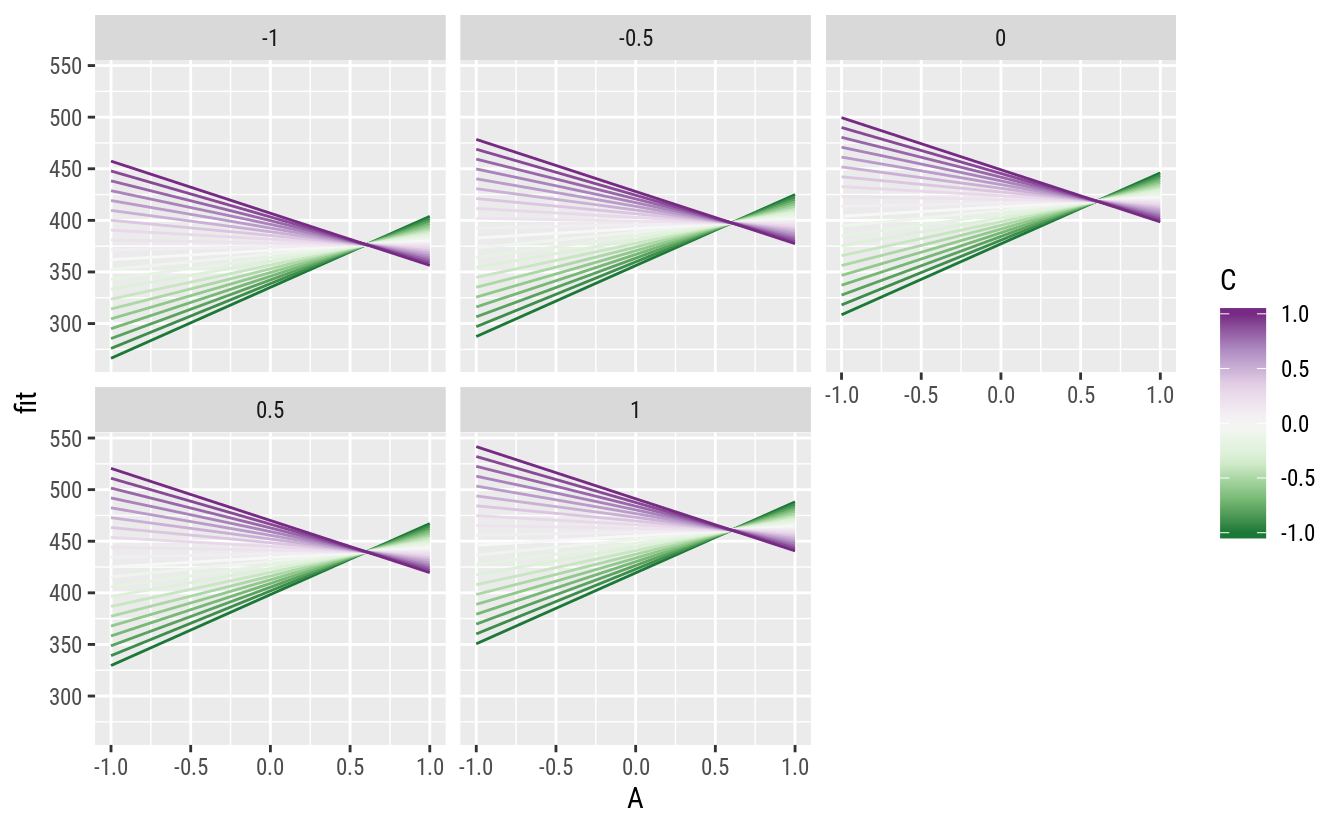
Quando \(C = 0\), praticamente não existe efeito de A. Quando \(C < 0\), o efeito de A é positivo. Quando \(C > 0\), o efeito de A é negativo. Como o efeito de B é aditivo aos demais, verifica-se que o seu efeito é positivo, ou seja, o aumento de B desloca a para cima o valor médio.
# Gráfico do valor para erro padrão em cada ponto.
ggplot(data = pred,
mapping = aes(x = A, y = C, z = se.fit, fill = se.fit)) +
facet_wrap(facets = ~B) +
geom_tile() +
scale_fill_distiller(palette = "RdBu") +
geom_contour(color = "black") +
coord_equal()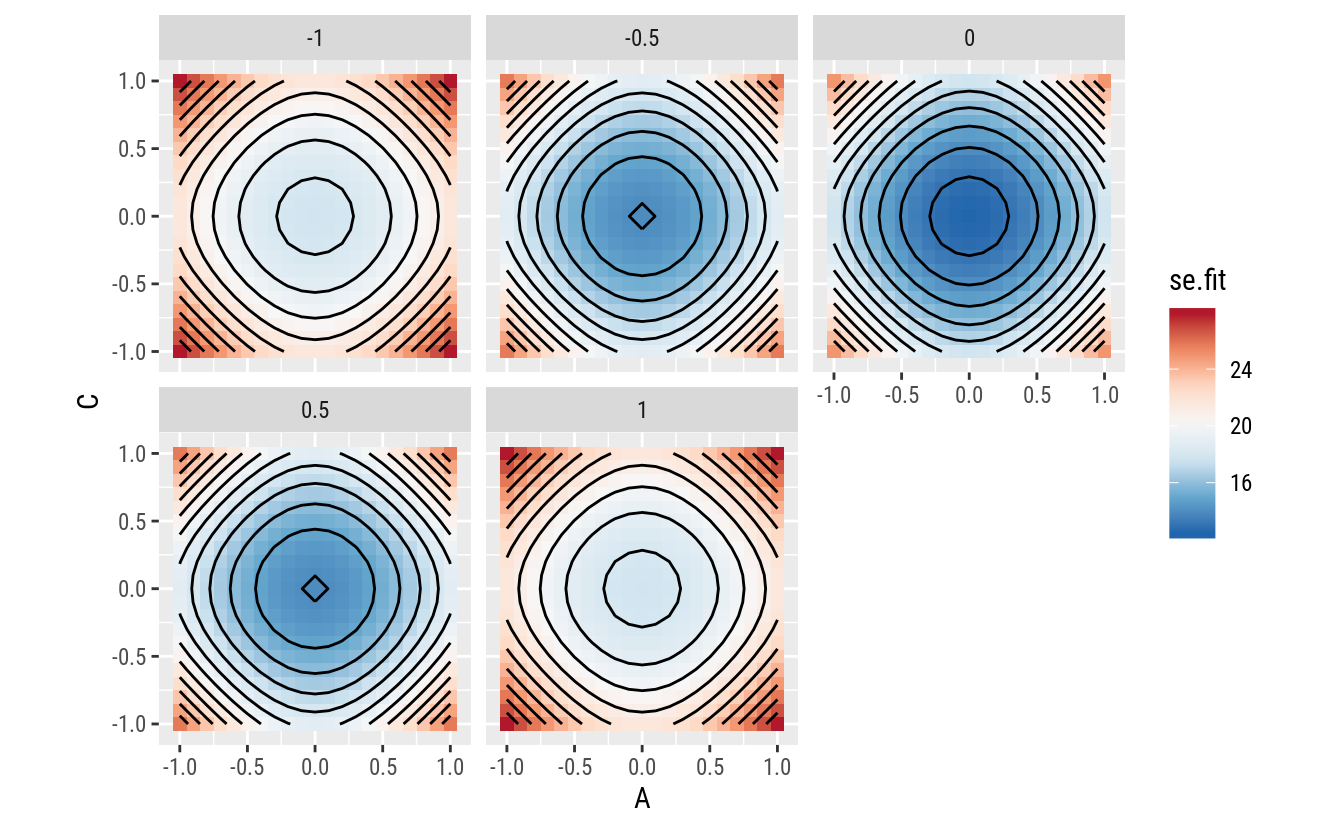
O erro padrão para a média aumenta a medida que o ponto experimental predito se afasta da origem da região experimental. No entanto, não depende da direção, apenas da distância. O comportamento é isotrópico, ou seja, os contornos são circulares. Essa é uma propriedade interessante de um delineamento. O erro padrão, para modelos em que a variância é constante, não depende do efeito dos fatores e sim apenas dos pontos de suporte do experimento.
No fragmento a seguir outros gráficos são confeccionados apenas para ilustrar outras formas de visualização. Eu particularmente não aconselho o uso de gráficos 3D.
# Esquema de cores para o gráfico com a `lattice`.
colr <- RColorBrewer::brewer.pal(11, "Spectral")
colr <- colorRampPalette(colr, space = "rgb")
# Gráfico de contornos de nível.
contourplot(fit ~ A + C | B,
data = pred,
cuts = 20,
aspect = 1,
as.table = TRUE,
region = TRUE,
col.regions = colr)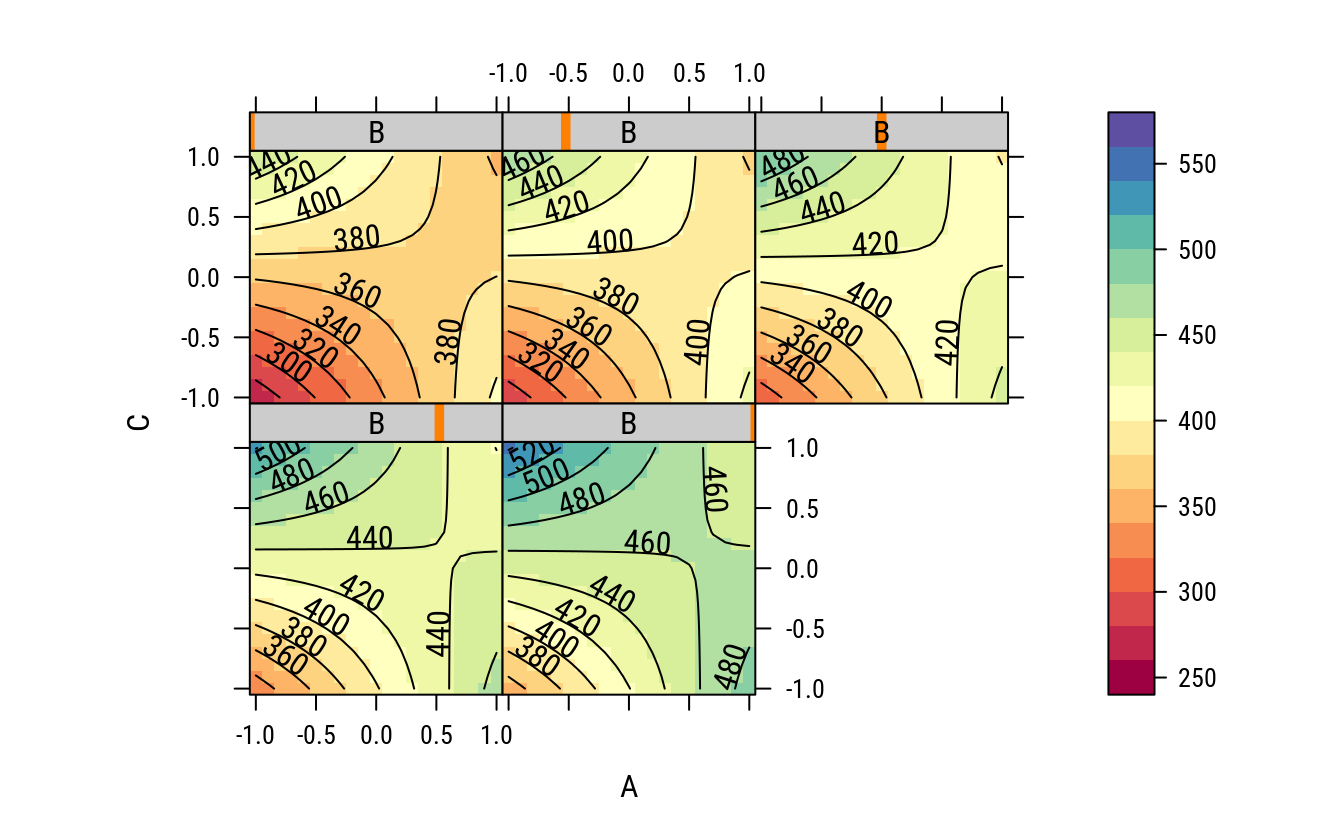
# Gráfico de superfície em 3D (não recomendo).
wireframe(fit ~ A + C | B,
data = pred,
as.table = TRUE,
drape = TRUE,
col.regions = colr(100))
# Apenas para mostrar que B desloca a superfície.
wireframe(fit ~ A + C, groups = B,
data = pred,
as.table = TRUE)
# Adicionando os contornos sobre a superfície. Requer funções externas.
source(paste0("https://raw.githubusercontent.com/walmes/wzRfun/",
"master/R/panel.3d.contour.R"))
# Gráfico de superfície em 3D com isolinhas (ainda não recomendo).
wireframe(fit ~ A + C | B,
data = pred,
as.table = TRUE,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100))
# Valores preditos em cada ponto experimental.
grid <- unique(ex14.12[, c("A", "B", "C")])
grid$fit <- predict(m1, newdata = grid)
arrange(grid, fit)## A B C fit
## 1 -1 -1 -1 266.375
## 2 -1 1 -1 350.625
## 3 1 -1 1 356.375
## 4 1 -1 -1 403.875
## 5 1 1 1 440.625
## 6 -1 -1 1 457.375
## 7 1 1 -1 488.125
## 8 -1 1 1 541.625Pela inspeção dos gráficos, a melhor combinação experimental no sentido de prolongar a vida da ferramente de corte é \[ \hat{y}_\text{opt} = f(A = -1, B = 1, C = 1) = 541.62. \]
Dado que o efeito de B é aditivo aos demais, a condicação de operação \(\{A = -1, C = 1\}\) é a melhor independente do nível de B, ou seja, independente da dureza do metal.
3.1.4 Próximo experimento
Pensando em otimizar a resposta, pode-se determinar a direção para a realização de um próximo experimento. Como B tem efeito aditivo, será determinada a direção considerando A e C apenas. Para isso precisa-se determinar o vetor gradiente.
# Termos da equação do modelo.
coef(m1)## (Intercept) B A C A:C
## 413.125 42.125 9.125 35.875 -59.625O vetor gradiente será determinado na origem do delineamento, ou seja, será a taxa na direção de cada fator. Determina-se isso com derivadas parciais avaliadas no ponto \(\{0, \ldots, 0\}\). A expressão do vertor gradiente está abaixo. \[ \begin{align*} \hat{y} &= f(x_1, \ldots, x_k; \hat{\beta}) = X \hat{\beta}\\ \nabla f(x_1, \ldots, x_k; \hat{\beta}) &= \left( \frac{\mathrm{d} f}{\mathrm{d} x_1}, \ldots, \frac{\mathrm{d} f}{\mathrm{d} x_k} \right).\\ \end{align*} \]
Como trata-se de um modelo linear, as derivadas são fáceis de determinar. Neste caso, o vetor é gradiente avaliado na origem do delineamento é \[\begin{align*} \nabla f(x_1, \ldots, x_k; \hat{\beta})\big |_{0,\ldots,0} &= \left( \frac{\mathrm{d} f}{\mathrm{d} x_1}, \ldots, \frac{\mathrm{d} f}{\mathrm{d} x_k} \right)\bigg |_{0,\ldots,0}\\ &= (\hat{\beta}_1, \ldots, \hat{\beta}_k). \end{align*}\]# Malha fina de valores para predição.
pred <- expand.grid(A = seq(-1, 1, by = 0.1),
C = seq(-1, 1, by = 0.1),
B = 0,
KEEP.OUT.ATTRS = FALSE)
X <- model.matrix(formula(m1)[-2], data = pred)
pred$fit <- c(X %*% coef(m1))
gg1 <-
ggplot(data = pred,
mapping = aes(x = A, y = C,
z = fit, fill = fit)) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1,
guide = FALSE) +
geom_contour(color = "black") +
geom_vline(xintercept = 0, linetype = 2) +
geom_hline(yintercept = 0, linetype = 2) +
coord_equal()
# Aproximação por um plano na origem do delineamento.
i <- c("(Intercept)", "A", "C")
pred$approx_grad <- c(X[, i] %*% coef(m1)[i])
gg2 <-
ggplot(data = pred,
mapping = aes(x = A, y = C,
z = approx_grad, fill = approx_grad)) +
geom_tile() +
scale_fill_distiller(palette = "Oranges",
direction = 1,
guide = FALSE) +
geom_contour(color = "black") +
coord_equal() +
geom_segment(data = data.frame(x = 0,
xend = coef(m1)["A"]/50,
y = 0,
yend = coef(m1)["C"]/50),
color = "white",
arrow = arrow(length = unit(0.2, "inches"),
angle = 30,
ends = "last"),
mapping = aes(x = x,
y = y,
xend = xend,
yend = yend,
z = NULL, fill = NULL))
gridExtra::grid.arrange(gg1, gg2, nrow = 1)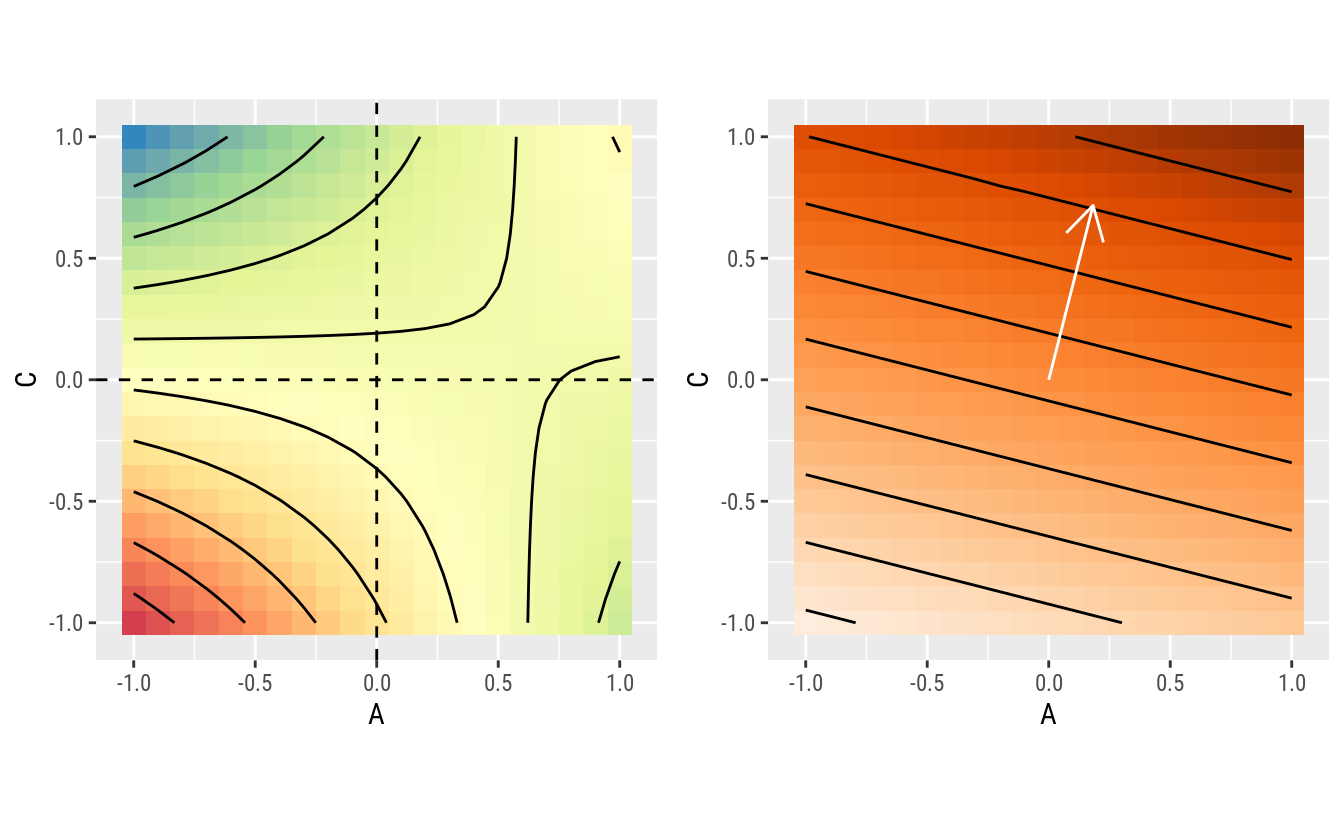
O gráfico acima mostra a direção indicada pelo vetor gradiente no ponto ${0, 0} de A e C. No entanto, faz mais sentido seguir o vetor gradiente no ponto \(\{-1, 1\}\) de A e C, já que esse é o ponto de máximo dentro da região experimental. Então agora precisa-se determinar o vetor gradiente no ponto \(\{-1, 1\}\), que será diferente daquele em \(\{0, 0\}\) já que existe interação.
\[ \begin{align*} \nabla f(x_1, x_3; \hat{\beta})\big |_{-1, 1} &= \left( \frac{\mathrm{d} f}{\mathrm{d} x_1}, \frac{\mathrm{d} f}{\mathrm{d} x_3} \right)\bigg |_{-1, 1}\\ &= \left( \frac{\mathrm{d} (\hat{\beta}_0 + \hat{\beta}_1 x_1 + \hat{\beta}_3 x_3 + \hat{\beta}_{13} x_1 x_3)}{ \mathrm{d} x_1}, \frac{\mathrm{d} (.))}{\mathrm{d} x_3} \right)\bigg |_{-1, 1}\\ &= (\hat{\beta}_1 + \hat{\beta}_{13}, \hat{\beta}_3 - \hat{\beta}_{13}). \end{align*} \]
# Malha fina de valores para predição.
pred <- expand.grid(A = seq(-3, 1, by = 0.1),
C = seq(-1, 3, by = 0.1),
B = 0,
KEEP.OUT.ATTRS = FALSE)
X <- model.matrix(formula(m1)[-2], data = pred)
pred$fit <- c(X %*% coef(m1))
design_square <- data.frame(x0 = -1, x1 = 1, y0 = -1, y1 = 1)
rect_design <- geom_rect(data = design_square,
inherit.aes = FALSE,
mapping = aes(xmin = x0, ymin = y0,
xmax = x1, ymax = y1),
color = "black",
fill = NA,
linetype = 2)
gg1 <-
ggplot(data = pred,
mapping = aes(x = A, y = C,
z = fit, fill = fit)) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1,
guide = FALSE) +
geom_contour(color = "black") +
rect_design +
coord_equal()
# Aproximação por um plano no ponto {-1, 1}.
i <- c("(Intercept)", "A", "C")
beta_loc <- rbind(coef(m1)["(Intercept)"],
coef(m1)["A"] + coef(m1)["A:C"],
coef(m1)["C"] - coef(m1)["A:C"])
pred$approx_grad <- c(X[, i] %*% beta_loc)
gg2 <-
ggplot(data = pred,
mapping = aes(x = A, y = C,
z = approx_grad, fill = approx_grad)) +
geom_tile() +
scale_fill_distiller(palette = "Oranges",
direction = 1,
guide = FALSE) +
geom_contour(color = "black") +
coord_equal() +
rect_design +
geom_segment(data = data.frame(x = -1,
xend = -1 + beta_loc[2]/50,
y = 1,
yend = 1 + beta_loc[3]/50),
color = "white",
arrow = arrow(length = unit(0.2, "inches"),
angle = 30,
ends = "last"),
mapping = aes(x = x,
y = y,
xend = xend,
yend = yend,
z = NULL, fill = NULL))
gridExtra::grid.arrange(gg1, gg2, nrow = 1)
Figura 3.1: Gráfico da superfície ajustada e da aproximação planar feita na origem do delineamento via vetor gradiente. A seta indica a direção para um próximo experimento. O código para esse gráfico foi omitido mas encontra-se disponível no código fonte.
Figura 3.2: Gráfico da superfície ajustada e da aproximação planar feita no ponto experimental \(\{-1, 1\}\) em A e C do delineamento via vetor gradiente. A seta indica a direção para um próximo experimento. O código para esse gráfico foi omitido mas encontra-se disponível no código fonte.
3.2 Sabor de um refrigerante
Imagina-se que 4 fatores influenciam o sabor de um refrigerante: tipo de adocante (A), razão de xarope e água (B), nível de carbonatação (C) e temperatura (D). Cada fator pode ser corrido em dois níveis, produzindo um planejamento \(2^4\). Em cada corrida do planejamento, amostras de refrigerante são dadas a 20 pessoas para testar. Cada pessoa atribui uma pontuação de 1 a 10 para o refrigerante. A pontuação total é a variável resposta avaliada e o objetivo é encontrar uma formulação que maximize a pontuação total. Duas réplicas desse planejamento são corridas e os resultados são mostrados na tabela.
Esses dados são do exerício 14-13, na página 354 do MONTGOMERY; RUNGER (2009).
3.2.1 Análise exploratória
Nessa seção serão feitos os cálculos para obter as estimativas dos parâmetros, somas de quadrados, predição e erros padrões. Na seção seguinte os dados serão analisados com funções do R e será feita discussão dos resultados.
rm(list = objects())
# Tabela contendo todos os pontos experimentais e repetições.
l <- c(-1, 1)
ex14.13 <- expand.grid(A = l,
B = l,
C = l,
D = l,
rept = 1:2,
KEEP.OUT.ATTRS = FALSE)
# Variável resposta do experimento.
ex14.13$y <- c(159, 168, 158, 166, 175, 179, 173, 179, 164, 187, 163,
185, 168, 197, 170, 194, 163, 175, 163, 168, 178, 183,
168, 182, 159, 189, 159, 191, 174, 199, 174, 198)
# Estrutura.
str(ex14.13)## 'data.frame': 32 obs. of 6 variables:
## $ A : num -1 1 -1 1 -1 1 -1 1 -1 1 ...
## $ B : num -1 -1 1 1 -1 -1 1 1 -1 -1 ...
## $ C : num -1 -1 -1 -1 1 1 1 1 -1 -1 ...
## $ D : num -1 -1 -1 -1 -1 -1 -1 -1 1 1 ...
## $ rept: int 1 1 1 1 1 1 1 1 1 1 ...
## $ y : num 159 168 158 166 175 179 173 179 164 187 ...# Repetições de cada ponto experimental.
ftable(xtabs(~A + B + C + D, data = ex14.13))## D -1 1
## A B C
## -1 -1 -1 2 2
## 1 2 2
## 1 -1 2 2
## 1 2 2
## 1 -1 -1 2 2
## 1 2 2
## 1 -1 2 2
## 1 2 2# Apenas um gráfico mas vários devem ser feitos.
ggplot(data = ex14.13,
mapping = aes(x = A, y = y, color = B)) +
facet_grid(facets = C ~ D) +
geom_point() +
stat_summary(mapping = aes(group = B),
fun.y = "mean",
geom = "line")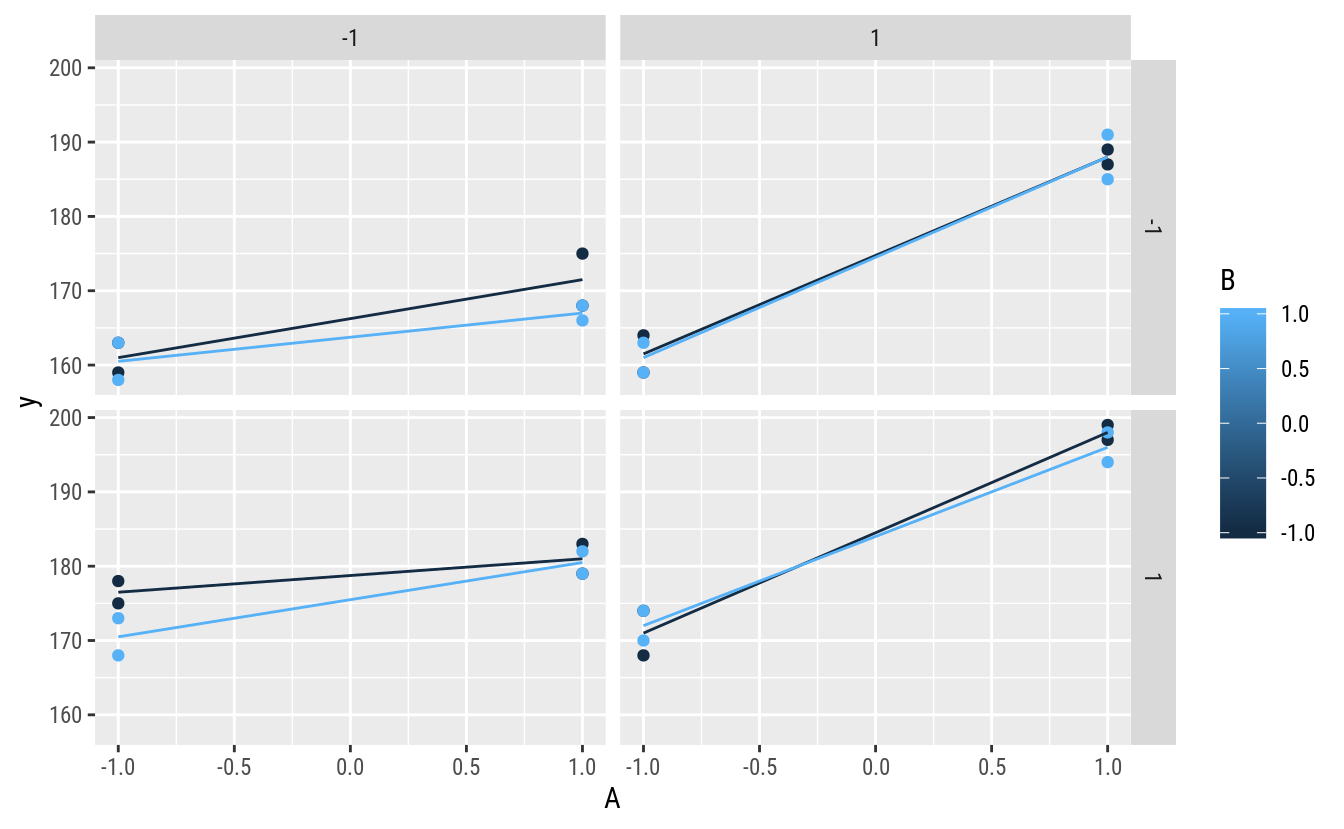
3.2.2 Análise feita de forma operacional
# Tabela de dados.
ex14.13## A B C D rept y
## 1 -1 -1 -1 -1 1 159
## 2 1 -1 -1 -1 1 168
## 3 -1 1 -1 -1 1 158
## 4 1 1 -1 -1 1 166
## 5 -1 -1 1 -1 1 175
## 6 1 -1 1 -1 1 179
## 7 -1 1 1 -1 1 173
## 8 1 1 1 -1 1 179
## 9 -1 -1 -1 1 1 164
## 10 1 -1 -1 1 1 187
## 11 -1 1 -1 1 1 163
## 12 1 1 -1 1 1 185
## 13 -1 -1 1 1 1 168
## 14 1 -1 1 1 1 197
## 15 -1 1 1 1 1 170
## 16 1 1 1 1 1 194
## 17 -1 -1 -1 -1 2 163
## 18 1 -1 -1 -1 2 175
## 19 -1 1 -1 -1 2 163
## 20 1 1 -1 -1 2 168
## 21 -1 -1 1 -1 2 178
## 22 1 -1 1 -1 2 183
## 23 -1 1 1 -1 2 168
## 24 1 1 1 -1 2 182
## 25 -1 -1 -1 1 2 159
## 26 1 -1 -1 1 2 189
## 27 -1 1 -1 1 2 159
## 28 1 1 -1 1 2 191
## 29 -1 -1 1 1 2 174
## 30 1 -1 1 1 2 199
## 31 -1 1 1 1 2 174
## 32 1 1 1 1 2 198# Matriz do modelo e vetor resposta.
# X <- model.matrix(~A * B * C * D, data = ex14.13)
X <- with(ex14.13,
cbind(I = 1,
A, B, C, D,
AB = A * B,
AC = A * C,
BC = B * C,
AD = A * D,
BD = B * D,
CD = C * D,
ABC = A * B * C,
ABD = A * B * D,
ACD = A * C * D,
BCD = B * C * D,
ABCD = A * B * C * D))
y <- cbind(ex14.13$y)
dim(X)## [1] 32 16# Matriz do modelo ao lado do vetor da resposta.
data.frame(X, "|" = "|", y, check.names = FALSE)## I A B C D AB AC BC AD BD CD ABC ABD ACD BCD ABCD | y
## 1 1 -1 -1 -1 -1 1 1 1 1 1 1 -1 -1 -1 -1 1 | 159
## 2 1 1 -1 -1 -1 -1 -1 1 -1 1 1 1 1 1 -1 -1 | 168
## 3 1 -1 1 -1 -1 -1 1 -1 1 -1 1 1 1 -1 1 -1 | 158
## 4 1 1 1 -1 -1 1 -1 -1 -1 -1 1 -1 -1 1 1 1 | 166
## 5 1 -1 -1 1 -1 1 -1 -1 1 1 -1 1 -1 1 1 -1 | 175
## 6 1 1 -1 1 -1 -1 1 -1 -1 1 -1 -1 1 -1 1 1 | 179
## 7 1 -1 1 1 -1 -1 -1 1 1 -1 -1 -1 1 1 -1 1 | 173
## 8 1 1 1 1 -1 1 1 1 -1 -1 -1 1 -1 -1 -1 -1 | 179
## 9 1 -1 -1 -1 1 1 1 1 -1 -1 -1 -1 1 1 1 -1 | 164
## 10 1 1 -1 -1 1 -1 -1 1 1 -1 -1 1 -1 -1 1 1 | 187
## 11 1 -1 1 -1 1 -1 1 -1 -1 1 -1 1 -1 1 -1 1 | 163
## 12 1 1 1 -1 1 1 -1 -1 1 1 -1 -1 1 -1 -1 -1 | 185
## 13 1 -1 -1 1 1 1 -1 -1 -1 -1 1 1 1 -1 -1 1 | 168
## 14 1 1 -1 1 1 -1 1 -1 1 -1 1 -1 -1 1 -1 -1 | 197
## 15 1 -1 1 1 1 -1 -1 1 -1 1 1 -1 -1 -1 1 -1 | 170
## 16 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 | 194
## 17 1 -1 -1 -1 -1 1 1 1 1 1 1 -1 -1 -1 -1 1 | 163
## 18 1 1 -1 -1 -1 -1 -1 1 -1 1 1 1 1 1 -1 -1 | 175
## 19 1 -1 1 -1 -1 -1 1 -1 1 -1 1 1 1 -1 1 -1 | 163
## 20 1 1 1 -1 -1 1 -1 -1 -1 -1 1 -1 -1 1 1 1 | 168
## 21 1 -1 -1 1 -1 1 -1 -1 1 1 -1 1 -1 1 1 -1 | 178
## 22 1 1 -1 1 -1 -1 1 -1 -1 1 -1 -1 1 -1 1 1 | 183
## 23 1 -1 1 1 -1 -1 -1 1 1 -1 -1 -1 1 1 -1 1 | 168
## 24 1 1 1 1 -1 1 1 1 -1 -1 -1 1 -1 -1 -1 -1 | 182
## 25 1 -1 -1 -1 1 1 1 1 -1 -1 -1 -1 1 1 1 -1 | 159
## 26 1 1 -1 -1 1 -1 -1 1 1 -1 -1 1 -1 -1 1 1 | 189
## 27 1 -1 1 -1 1 -1 1 -1 -1 1 -1 1 -1 1 -1 1 | 159
## 28 1 1 1 -1 1 1 -1 -1 1 1 -1 -1 1 -1 -1 -1 | 191
## 29 1 -1 -1 1 1 1 -1 -1 -1 -1 1 1 1 -1 -1 1 | 174
## 30 1 1 -1 1 1 -1 1 -1 1 -1 1 -1 -1 1 -1 -1 | 199
## 31 1 -1 1 1 1 -1 -1 1 -1 1 1 -1 -1 -1 1 -1 | 174
## 32 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 | 198k <- 4 # Número de fatores.
r <- 2 # Número de repetições.
# Contrates são o principal artefato. É apenas X'y.
ctr <- crossprod(X, y)
ctr## [,1]
## I 5608
## A 272
## B -26
## C 174
## D 134
## AB -2
## AC -10
## BC -4
## AD 146
## BD 20
## CD -20
## ABC 12
## ABD -8
## ACD 0
## BCD 2
## ABCD -26# Estimativas dos parâmetros.
# beta <- solve(crossprod(X), crossprod(X, y))
beta <- diag(1/(r * 2^k), 2^k) %*% ctr
beta## [,1]
## [1,] 175.2500
## [2,] 8.5000
## [3,] -0.8125
## [4,] 5.4375
## [5,] 4.1875
## [6,] -0.0625
## [7,] -0.3125
## [8,] -0.1250
## [9,] 4.5625
## [10,] 0.6250
## [11,] -0.6250
## [12,] 0.3750
## [13,] -0.2500
## [14,] 0.0000
## [15,] 0.0625
## [16,] -0.8125# Estimativa dos parâmetros (por somatórios basicamente).
ctr/(r * 2^k) # idem ao `beta`## [,1]
## I 175.2500
## A 8.5000
## B -0.8125
## C 5.4375
## D 4.1875
## AB -0.0625
## AC -0.3125
## BC -0.1250
## AD 4.5625
## BD 0.6250
## CD -0.6250
## ABC 0.3750
## ABD -0.2500
## ACD 0.0000
## BCD 0.0625
## ABCD -0.8125# Somas de quadrados.
tail(ctr, n = -1)^2/(r * 2^k)## [,1]
## A 2312.000
## B 21.125
## C 946.125
## D 561.125
## AB 0.125
## AC 3.125
## BC 0.500
## AD 666.125
## BD 12.500
## CD 12.500
## ABC 4.500
## ABD 2.000
## ACD 0.000
## BCD 0.125
## ABCD 21.125# Valores ajustados para cada ponto experimental.
ex14.13$hy <- X %*% beta
# Resíduos.
ex14.13$res <- with(ex14.13, y - hy)
# Desvio padrão residual.
s2 <- with(ex14.13, sum(res^2)/(nrow(X) - ncol(X)))
s2## [1] 9.5625# Erros padrões das estimativas dos parâmetros.
# summary(m0)$coefficients
sqrt(s2/(r * 2^k))## [1] 0.5466517# Erros padrões das médias nos pontos experimentais.
# predict(m0, se.fit = TRUE)$se.fit
sqrt(s2/r)## [1] 2.186607# Matriz para a predição de um ponto qualquer.
# Xnew <- model.matrix(~A * B * C,
# data = data.frame(A = -1, B = 0.25, C = 0.5))
Xnew <- with(data.frame(A = -0.5, B = 0.9, C = 0.5, D = 0),
cbind(I = 1,
A, B, C, D,
AB = A * B,
AC = A * C,
BC = B * C,
AD = A * D,
BD = B * D,
CD = C * D,
ABC = A * B * C,
ABD = A * B * D,
ACD = A * C * D,
BCD = B * C * D,
ABCD = A * B * C * D))
Xnew## I A B C D AB AC BC AD BD CD ABC ABD ACD BCD
## [1,] 1 -0.5 0.9 0.5 0 -0.45 -0.25 0.45 0 0 0 -0.225 0 0 0
## ABCD
## [1,] 0# Predição em um ponto qualquer.
Xnew %*% beta## [,1]
## [1,] 172.9531# Variância da predição em um ponto qualquer.
# Xnew %*% (s2 * solve(crossprod(X))) %*% t(Xnew)
Xnew %*% (s2 * diag(1/(r * 2^k), 2^k)) %*% t(Xnew)## [,1]
## [1,] 0.84512333.2.3 Análise feita com funções
Nessa sessão serão usadas as funções do R para análise dos dados. Como é um modelo linear, usa-se a lm().
# Ajuste do modelo saturado.
m0 <- lm(y ~ A * B * C * D, data = ex14.13)
# Diagnóstico nos resíduos.
par(mfrow = c(1, 2))
plot(m0, which = 3)
plot(m0, which = 2)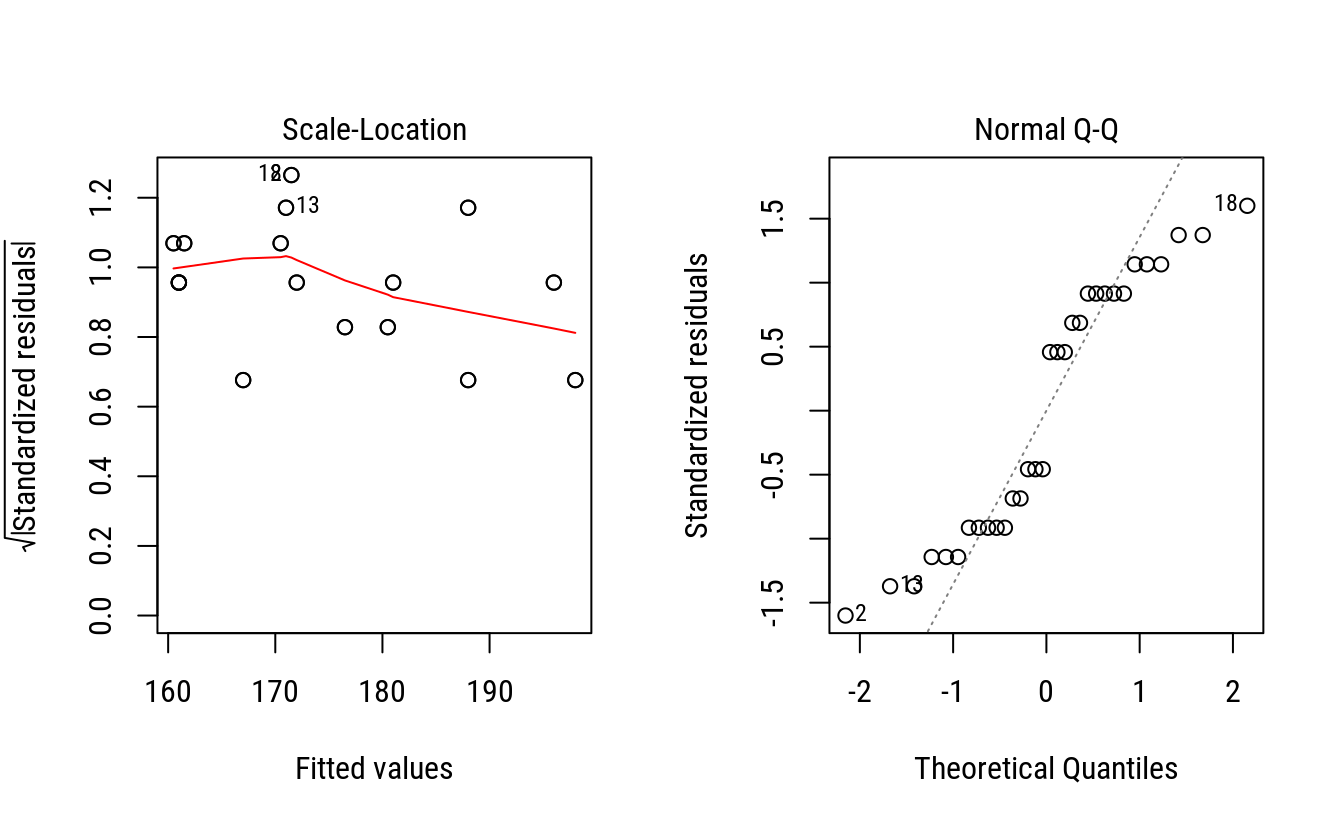
layout(1)Pela inspeção dos gráficos, não existem evidências contra o atendimento dos pressupostos. Obviamente que por serem poucas observações, dificulta-se detectar padrões característicos, como relação média-variância. O que pode ser comentado é que o fato da variável ser medida em escala discreta (soma de 20 termos com notas de 0 a 10), isso aparece no gráfico com resíduos padronizados de mesmo valor.
# Quadro de análise de variância.
anova(m0)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## A 1 2312.00 2312.00 241.7778 4.451e-11 ***
## B 1 21.12 21.12 2.2092 0.1566
## C 1 946.13 946.13 98.9412 2.958e-08 ***
## D 1 561.13 561.13 58.6797 9.692e-07 ***
## A:B 1 0.13 0.13 0.0131 0.9104
## A:C 1 3.13 3.13 0.3268 0.5755
## B:C 1 0.50 0.50 0.0523 0.8220
## A:D 1 666.12 666.12 69.6601 3.187e-07 ***
## B:D 1 12.50 12.50 1.3072 0.2697
## C:D 1 12.50 12.50 1.3072 0.2697
## A:B:C 1 4.50 4.50 0.4706 0.5025
## A:B:D 1 2.00 2.00 0.2092 0.6536
## A:C:D 1 0.00 0.00 0.0000 1.0000
## B:C:D 1 0.13 0.13 0.0131 0.9104
## A:B:C:D 1 21.13 21.13 2.2092 0.1566
## Residuals 16 153.00 9.56
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Estimativas.
summary(m0)##
## Call:
## lm(formula = y ~ A * B * C * D, data = ex14.13)
##
## Residuals:
## Min 1Q Median 3Q Max
## -3.5 -2.0 0.0 2.0 3.5
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 1.752e+02 5.467e-01 320.588 < 2e-16 ***
## A 8.500e+00 5.467e-01 15.549 4.45e-11 ***
## B -8.125e-01 5.467e-01 -1.486 0.157
## C 5.438e+00 5.467e-01 9.947 2.96e-08 ***
## D 4.188e+00 5.467e-01 7.660 9.69e-07 ***
## A:B -6.250e-02 5.467e-01 -0.114 0.910
## A:C -3.125e-01 5.467e-01 -0.572 0.575
## B:C -1.250e-01 5.467e-01 -0.229 0.822
## A:D 4.562e+00 5.467e-01 8.346 3.19e-07 ***
## B:D 6.250e-01 5.467e-01 1.143 0.270
## C:D -6.250e-01 5.467e-01 -1.143 0.270
## A:B:C 3.750e-01 5.467e-01 0.686 0.503
## A:B:D -2.500e-01 5.467e-01 -0.457 0.654
## A:C:D 1.305e-14 5.467e-01 0.000 1.000
## B:C:D 6.250e-02 5.467e-01 0.114 0.910
## A:B:C:D -8.125e-01 5.467e-01 -1.486 0.157
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 3.092 on 16 degrees of freedom
## Multiple R-squared: 0.9676, Adjusted R-squared: 0.9371
## F-statistic: 31.81 on 15 and 16 DF, p-value: 4.504e-09Pela avaliação do quadro de análise de variância, não houve interações relevantes envolvendo C (nível de carbonação), apenas o efeito principal. Não houve efeito de B (xarope/água) em nenhum termo. Houve interação dupla entre A (adoçante) e D (temperatura). Portanto, alguns termos do modelo podem ser abandonados.
# Ajuste do modelo reduzido apenas nos termos relevantes.
m1 <- update(m0, . ~ C + A * D)
anova(m1)## Analysis of Variance Table
##
## Response: y
## Df Sum Sq Mean Sq F value Pr(>F)
## C 1 946.13 946.13 110.766 4.688e-11 ***
## A 1 2312.00 2312.00 270.673 1.347e-15 ***
## D 1 561.13 561.13 65.693 1.046e-08 ***
## A:D 1 666.12 666.12 77.985 1.905e-09 ***
## Residuals 27 230.63 8.54
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# Variâncias residuais.
c(s2_m0 = summary(m0)$sigma^2,
s2_m1 = summary(m1)$sigma^2)## s2_m0 s2_m1
## 9.562500 8.541667# Teste para nulidade dos termos abandonados.
anova(m1, m0)## Analysis of Variance Table
##
## Model 1: y ~ C + A + D + A:D
## Model 2: y ~ A * B * C * D
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 27 230.62
## 2 16 153.00 11 77.625 0.738 0.6907O modelo reduzido ajustado pelo abandono daqueles não relevantes não diferiu, conforme esperado, do modelo maximal. Neste exemplo, a variância residual do modelo m1 foi menor que do modelo m0. Todavia, a diferença nessas estimativas é irrelevante. Poderia-se usar a estimativa pura da variância residual fornecida pelo modelo m0. Os resultados não devem mudar substancialmente.
# Malha fina de valores para predição.
pred <- expand.grid(A = seq(-1, 1, by = 0.1),
C = seq(-1, 1, by = 0.5),
D = seq(-1, 1, by = 0.1),
KEEP.OUT.ATTRS = FALSE)
pred <- cbind(pred,
as.data.frame(predict(m1,
newdata = pred,
se.fit = TRUE)[1:2]))
# Gráfico da superfície média (valores ajustados).
ggplot(data = pred,
mapping = aes(x = A, y = D, z = fit, fill = fit)) +
facet_wrap(facets = ~C) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1) +
geom_contour(color = "black") +
coord_equal()
A função apresenta um comportamento que, conforme antecipado, indica que o efeito de A (adoçante) depende do valor de D (temperatura) e vice versa. É possível entender melhor com gráfico de linhas.
# Gráficos de linhas.
ggplot(data = pred,
mapping = aes(x = A, y = fit, color = D, group = D)) +
facet_wrap(facets = ~C) +
geom_line() +
scale_color_distiller(palette = "PRGn")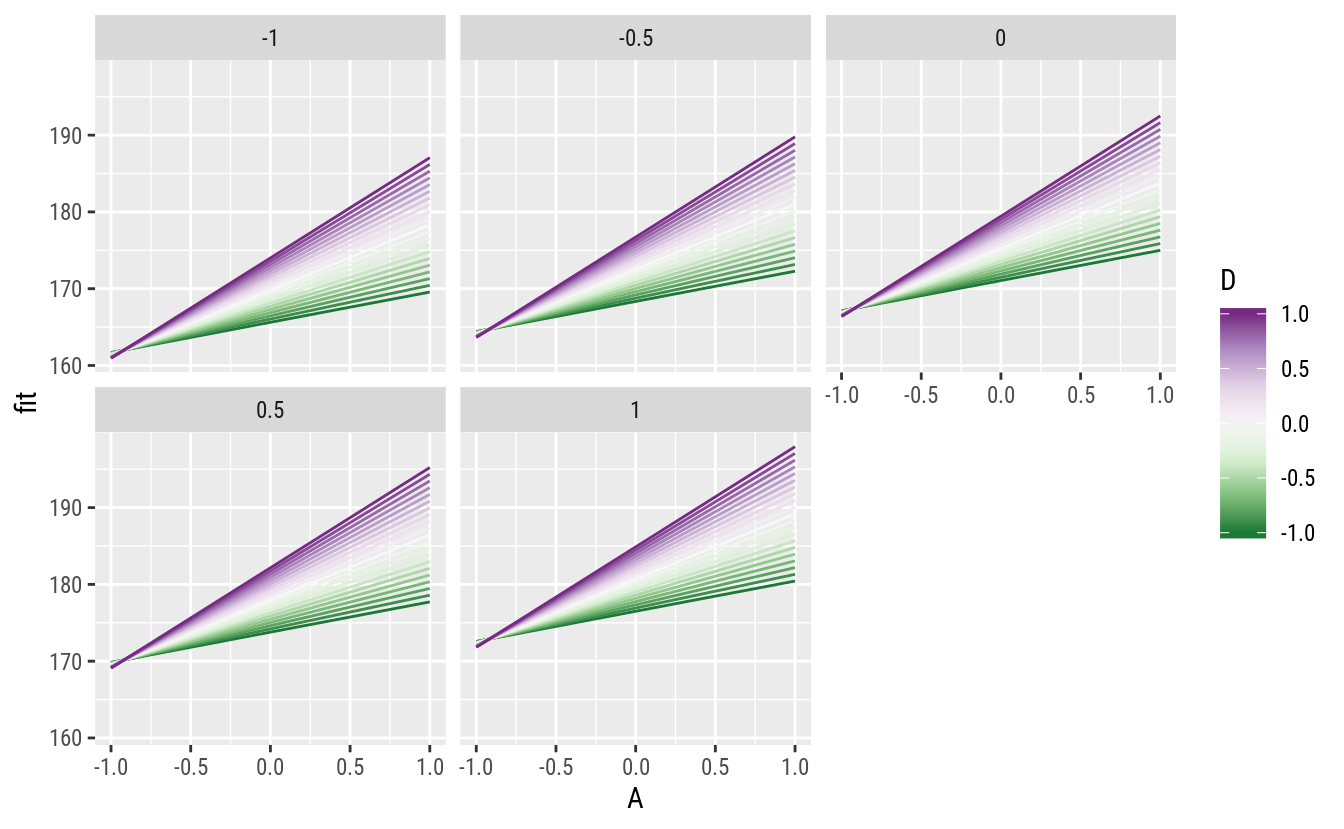
Verifica-se que quanto maior o valor de D (temperatura), mais pronunciado é o efeito de A (adoçante). Diz-se que existe sinergismo entre esses fatores. O efeito de C (carbonação) é aditivo aos demais (nas escalas consideradas). Quanto maior C, maior o valor médio fixando-se os demais fatores.
No fragmento a seguir outros gráficos são confeccionados apenas para ilustrar outras formas de visualização. Eu particularmente não aconselho o uso de gráficos 3D.
# Esquema de cores para o gráfico com a `lattice`.
colr <- RColorBrewer::brewer.pal(11, "Spectral")
colr <- colorRampPalette(colr, space = "rgb")
# Adicionando os contornos sobre a superfície. Requer funções externas.
source(paste0("https://raw.githubusercontent.com/walmes/wzRfun/",
"master/R/panel.3d.contour.R"))
# Gráfico de superfície em 3D com isolinhas (ainda não recomendo).
wireframe(fit ~ A + D | C,
data = pred,
as.table = TRUE,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100))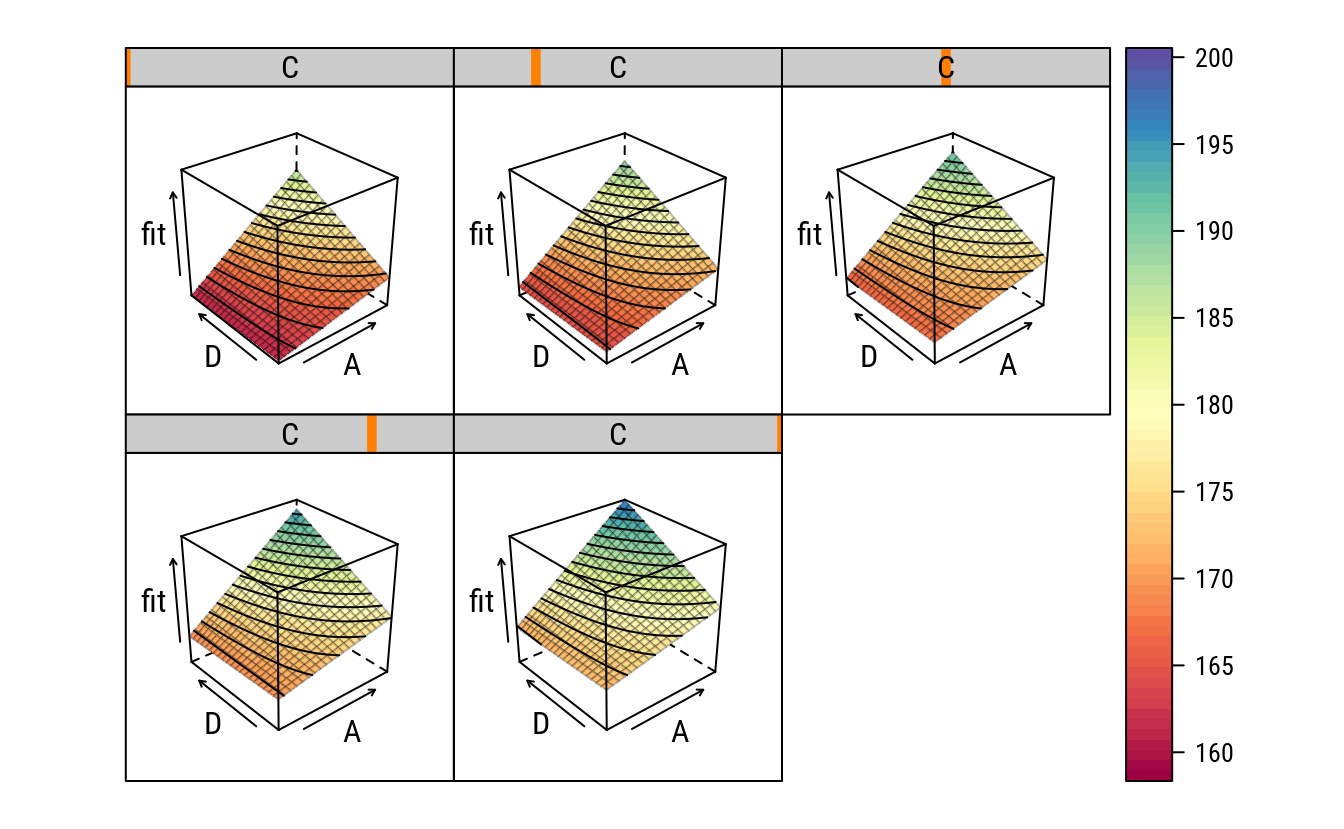
# Valores preditos em cada ponto experimental.
grid <- unique(ex14.13[, c("A", "C", "D")])
grid$fit <- predict(m1, newdata = grid)
arrange(grid, fit)## A C D fit
## 1 -1 -1 1 160.9375
## 2 -1 -1 -1 161.6875
## 3 1 -1 -1 169.5625
## 4 -1 1 1 171.8125
## 5 -1 1 -1 172.5625
## 6 1 1 -1 180.4375
## 7 1 -1 1 187.0625
## 8 1 1 1 197.9375Pela inspeção dos gráficos, a melhor combinação experimental no sentido de aumentar a nota para o refrigerante é \[ \hat{y}_\text{opt} = f(A = 1, C = 1, D = 1) = 197.94. \]
Dado que o efeito de C (carbonação) é aditivo aos demais, a condicação de operação \(\{A = 1, D = 1\}\) é a melhor independente do nível de C, ou seja, independente da carbonação.
3.2.4 Próximo experimento
Seguindo mesmo procedimento da sessão anterior, o gráfico abaixo indica a direção para um próximo experimento, considerando os fatores A e D. O vetor gradiente é determinado no ponto \(\{1, 1\}\) de A e D.
# Malha fina de valores para predição.
pred <- expand.grid(A = seq(-1, 3, by = 0.1),
D = seq(-1, 3, by = 0.1),
C = 0,
KEEP.OUT.ATTRS = FALSE)
X <- model.matrix(formula(m1)[-2], data = pred)
pred$fit <- c(X %*% coef(m1))
design_square <- data.frame(x0 = -1, x1 = 1, y0 = -1, y1 = 1)
rect_design <- geom_rect(data = design_square,
inherit.aes = FALSE,
mapping = aes(xmin = x0, ymin = y0,
xmax = x1, ymax = y1),
color = "black",
fill = NA,
linetype = 2)
gg1 <-
ggplot(data = pred,
mapping = aes(x = A, y = D,
z = fit, fill = fit)) +
geom_tile() +
scale_fill_distiller(palette = "Spectral",
direction = 1,
guide = FALSE) +
geom_contour(color = "black") +
rect_design +
coord_equal()
# Aproximação por um plano no ponto {-1, 1}.
i <- c("(Intercept)", "A", "D")
beta_loc <- rbind(coef(m1)["(Intercept)"],
coef(m1)["A"] + coef(m1)["A:D"],
coef(m1)["D"] + coef(m1)["A:D"])
pred$approx_grad <- c(X[, i] %*% beta_loc)
gg2 <-
ggplot(data = pred,
mapping = aes(x = A, y = D,
z = approx_grad, fill = approx_grad)) +
geom_tile() +
scale_fill_distiller(palette = "Oranges",
direction = 1,
guide = FALSE) +
geom_contour(color = "black") +
coord_equal() +
rect_design +
geom_segment(data = data.frame(x = 1,
xend = 1 + beta_loc[2]/10,
y = 1,
yend = 1 + beta_loc[3]/10),
color = "white",
arrow = arrow(length = unit(0.2, "inches"),
angle = 30,
ends = "last"),
mapping = aes(x = x,
y = y,
xend = xend,
yend = yend,
z = NULL, fill = NULL))
gridExtra::grid.arrange(gg1, gg2, nrow = 1)
# Empilha.
predw <- pred %>%
gather(key = "surface", value = "value", fit, approx_grad)
# Gráfico de superfície mostrando as duas.
wireframe(value ~ A + D | surface,
drape = TRUE,
col = rgb(0, 0, 0, 0.25),
panel.3d.wireframe = panel.3d.contour,
col.contour = 1,
type = "on",
col.regions = colr(100),
data = predw)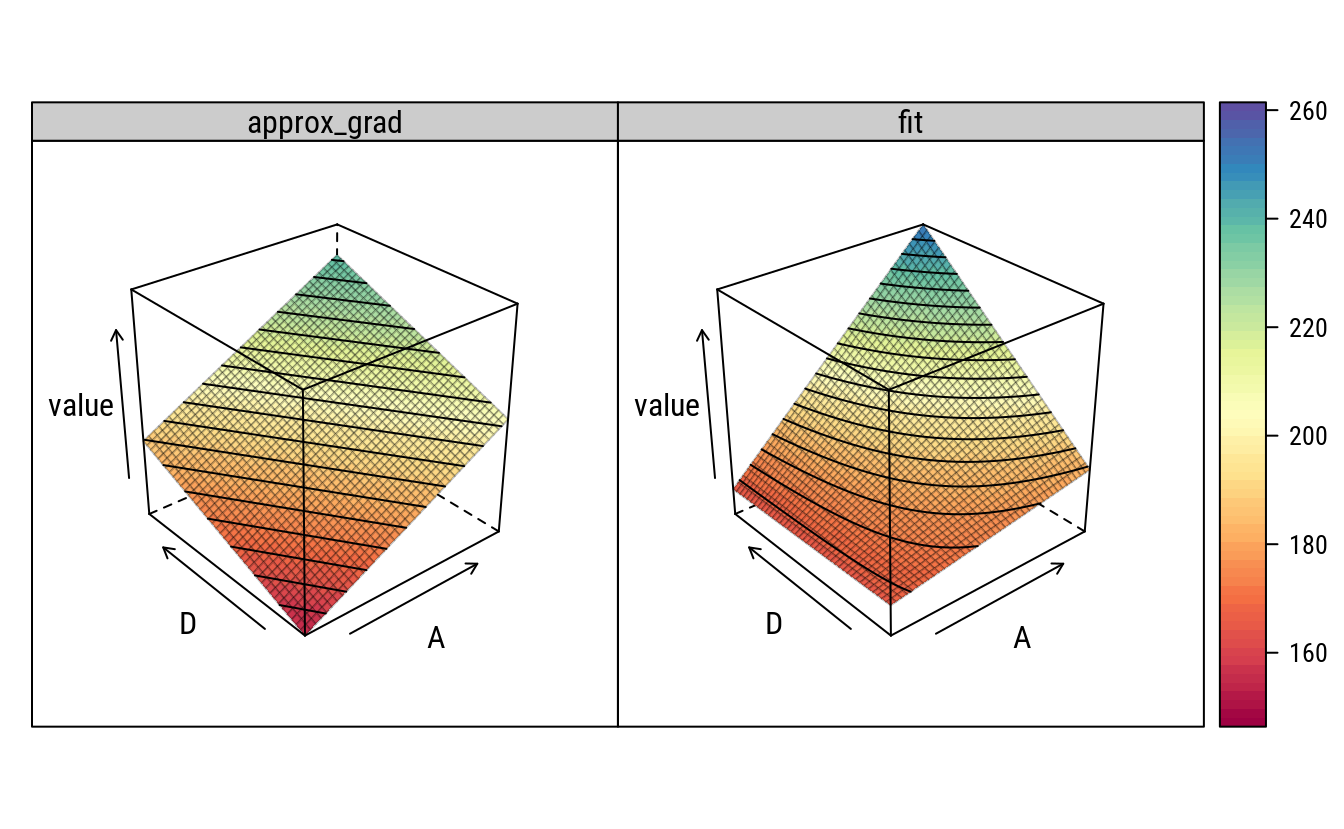
Referências Bibliográficas
MONTGOMERY, D. C. Design and analysis of experiments. 5th ed. New York: John Wiley & Sons, 2001.
MONTGOMERY, D. C.; RUNGER, G. C. Estatística aplicada e probabilidade para engenheiros. Rio de Janeiro: Livros Técnicos e Científicos, 2009.